(Ciencias de Joseleg) (Biología) (Teoría
de la Biología)
(Genética moderna) (Introducción) (Naturaleza
química del gen) (Conflicto
por la estructura del ADN) (Estructura
del ADN) (La
replicación del ADN) (Síntesis
de proteínas) (Denaturación
y renaturación) (Estructura
del genoma) (Tipos
de mutaciones) (Elementos
móviles del genoma) (Identificación
humana) (Referencias
bibliográficas)
Evolución
de los genes de la globina
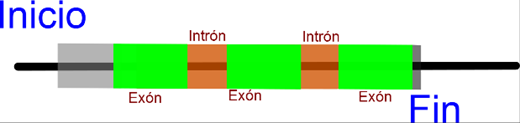
La hemoglobina humana es un sistema relativamente complejo con cuatro unidades integradas, estas unidades son proteínas en sí mismas con una estructura terciara bien determinada. Por esta razón la hemoglobina humana se denomina una proteína con estructura cuaternaria. Cada unidad será denominada polipéptido y la hemoglobina humana posee cuatro. La estructura de cada globina cuando se analiza su gen productor siempre posee la misma estructura, tres exones y dos intrones. ¿Qué es un exón y que es un intrón? Bueno a pesar de que cubrimos parte de la estructura del genoma eucariota aun no los hemos mencionado.
Figura 69. Maduración del mensajero de ARN. Procesamiento del ARN mensajero, inicialmente
cuando las enzimas crean el ARN lo leen como una sola entidad, pero
posteriormente otras enzimas editan o maduran al ARN mensajero, transformándolo
en el ARN mensajero maduro sin intrones.
Los genes no son exactamente como los simbolizamos. En la figura 1 se muestra el símbolo estándar para un gen, donde la barra gruesa a color simboliza el gen, mientras que la barra delgada simboliza el ADN no codificante. En realidad, la estructura se asemejaría más a esto (Figura 69) en la cual los genes se encuentran divididos por secuencias de ADN no codificante.
Figura
70. Estructura del gen
de la globina.
Cuando las enzimas especializadas transforman el ADN a el ARN mensajero hay un paso de edición en la cual los intrones son editados del mensaje, quedando de esta forma un mensaje compuesto solo por algunos fragmentos originales. El fragmento de ADN que es eliminado por la edición intermedia en el mensajero de ADN se denomina intrón, mientras que la parte del mensaje que si es enviada al ribosoma para convertirse en proteínas se denomina exón.
Figura 71. Estructura del gen
de la globina.
Como se
mencionó anteriormente, aun cuando los genes al neofuncionalizar y
subfuncionalizar alteran parte de su estructura, la mayor parte continua
intacta, lo cual le permite a los biólogos buscar en los diferentes seres vivos
miembros de una familia de genes determinada, y dependiendo de la cantidad de
pares de bases que se asemejen pueden inferir el parentesco. Entre más antigua
fue la separación de los genes más mutaciones han acumulado y por lo tanto más
diferentes serán.
Cuando
los científicos realizaron la búsqueda por las globinas se encontraron miembros
de la familia realizando funciones en otros contextos de los animales, como por
ejemplo las mioglobinas, pero también encontraron miembros de la familia en las
plantas, ya que estas producen una proteína llamada le hemoglobina
"portador de nitrógeno". En otros los casos no solo la secuencia en
sí, sino la estructura misma del gen con tres exones dos intrones se mantenían
constante, como el apellido de un clan.
Esto
nos revela varias cosas, en primera instancia, las globinas como tal realizan
funciones relacionadas con el transporte de electrones y la captura de oxígeno,
dióxido de carbono y monóxido de carbono en diferentes contextos, y que como
unidades independientes son completamente funcionales, lo cual va en contra del
concepto de complejidad irreductible.
Si
debemos creerle a Darwin, el origen de la hemoglobina debe encontrarse en un
monómero, un solo fragmento que cumpla con la función. Evidentemente no podemos
buscar en el sistema biológico humano debido a que en ese contexto se necesita
la forma tetramérica, después de todo tanto el sistema como sus componentes
evolucionan simultáneamente. Por lo anterior y debido a que las moléculas no se
fosilizan se debe emplear la genética comparada. En este caso lo más objetivo
es buscar en los familiares más lejanos, en el caso de los vertebrados,
aquellos peces que se separaron del resto de los linajes antes de que
evolucionaran las vértebras o las mandíbulas. Estos peces primitivos son los
mixinos y las lampreas.
Al
buscar en estos peces se encontró “oh sorpresa” que la hipótesis darwiniana
queda soportada, en estos peces solo hay una copia del gen de la globina, lo
que sugiere entre otras que los genes de la globina aparecieron por duplicación
y que estos peses se separaron del resto antes de que esa duplicación diera
lugar.
Con la evolución de los peces vertebrados en algún punto el gen de la globina se duplicó, terminando la copia de respaldo cerca de la copia original en el mismo cromosoma. Sin embargo, los loci de las globinas son muy móviles y pueden trasladarse entre cromosomas. Por ejemplo, mientras que en los peces cebra y el las ranas del género Xenopus spp., ambos genes se encuentran en loci adyacentes. Por el contrario, en los vertebrados posteriores, uno de los genes de la globina saltó a otro cromosoma. Posteriormente, cada gen llevó a cabo una serie de duplicaciones y reorganizaciones generando los diversos tipos de globinas de los mamíferos actuales.
Figura
72. Esquema de la evolución de la familia de
genes de la globina en los vertebrados. (1)
simplificación de una globina ancestral, (2,3) duplicación del locus de la
globina, (4,5) separación de los loci de las globinas en cromosomas
independientes, (6,7) duplicaciones y reorganizaciones posteriores de los loci
de las globinas.
La
hemoglobina del adulto está compuesta por cuatro polipéptidos de dos tipos, una
alfa y la otra beta, pero esa no es toda la historia. Esto se debe a que los
seres humanos poseemos otras copias del gen de la globina que permiten la
producción de otros tipos de hemoglobina durante el desarrollo fetal. En el
cromosoma 11 se encuentra la subfamilia de globinas beta, la cual se divide en
globinas beta, gama y épsilon. Específicamente los loci son: globina beta, la globina delta, el pseudogen psi-beta,
globina fetal gama A, globina fetal gama B, y globina embrionaria épsilon. En
el cromosoma 16 se encuentra la subfamilia alfa, la cual se divide en las
globinas alfa y zeta. Específicamente los
loci
son: globina alfa 1, globina alfa 2, pseudogen psi-alfa 1, pseudo gen psi-zeta
y globina embrionaria zeta.
Esta
familia de genes ilustra perfectamente el concepto de subfuncionalización
mediante la especialización funcional. Cada una de las globinas de la familia
alfa puede unirse con alguna de las beta, la hemoglobina resultante posee
funciones específicas dependiendo del sistema en que se encuentre, sea este un
embrión un feto o un adulto.
En los
nidos “clusters” de las globinas aparte de encontrar los genes de las cadenas
extra que ya habían sido identificadas años atrás, los genetistas también se
encontraron con
Dicho
de otra forma, un pseudogen es lo que queda de un gen antiguo que acumuló SNP
de forma tal que en lugar de adquirir una función o especializarse perdió toda
su funcionalidad. Tanto en el cromosoma
11 como en el 16 hay
El
genoma humano posee 19 000 loci de pseudogenes, lo cual
contrasta con los 20 500 loci para genes funcionales, es
decir, casi la mitad de los
Una de
las características más sospechosas de las globinas es que en comparación con
otros genes poseen una cantidad sorprendentemente pequeña de ADN no
codificante, en otras palabras, el gen posee una integridad distintiva sin
importar cuantas veces se hubiera copiado. Para repasar, las globinas parecen
poder saltar de un lugar a otro manteniendo su estructura básica de forma
intacta. O lo que es lo mismo, es como si el gen pudiera copiarse y viajar a
otro cromosoma e insertarse de forma integral. El mecanismo que permite esta proeza
genética es lo que permitió formular el concepto de genes saltarines o
trasposones.
Si bien
una parte integral de las explicaciones evolutivas recurren al azar, no todas
las respuestas deben o pueden recurrir al azar debido a las probabilidades tan
bajas. Por ejemplo, la probabilidad de que un
Figura
73. Barbara McClintock. Es la madre de la
teoría de los genes saltarines (YouTube; YouTube), concepto que ha
dado claridad a muchos procesos en
genética evolutiva, y aplicaciones médicas como en el cáncer.
La hipótesis de los genes saltarines surgió como una forma de explicar cómo es que ocurrían genes duplicados en cromosomas diferentes, sin que estos tuvieran evidencia de una duplicación cromosómica mayor. Si se tratara de una poliploidía todo el cromosoma sería una copia homóloga de otro cromosoma al interior del mismo genoma, pero en estos casos lo único que es homólogo es una leve fracción, un locus con un cromosoma duplicado. La primera persona en sugerir que estos elementos se podían trasladar de un lugar al otro del genoma, es decir que podían saltar fue Barbara McClintock “1902-1992” trabajando en el laboratorio de Harbor en New York. El modelo biológico que permitió el desarrollo de este modelo fue el maíz.
Figura
74. Mecanismo de transposición. La transposición es el movimiento de un elemento
móvil, el cual puede estar compuesto por varios loci así como elementos
reguladores, de un lugar del genoma a otro
Los
rasgos genéticos del maíz pueden revelarse en cambios de sus patrones en las
hojas y en los patrones de coloración de las semillas. A finales de la década
de los 40s Mc-Clintock encontró un grupo de mutaciones muy inestable que
aparecían y desaparecían de una generación a la siguiente o inclusive durante
la historia de vida de un solo individuo. Después de muchos años de estudio,
ella concluyó que ciertos elementos genéticos pueden moverse de un cromosoma a
otro. A estos elementos móviles ella los denominó Transponibles y a su
organización transposón. Sin embargo, en la actualidad se usa el término
transposón para designar al elemento móvil del ADN.
Simultáneamente,
otros biólogos moleculares trabajando en bacterias no pudieron encontrar
evidencias de estos elementos móviles. En sus estudios, los genes aparentaban
ser elementos estables organizados en patrones lineares al interior del
cromosoma circular, sin la presencia de secuencias no codificantes o
pseudogenes. Los resultados de McClintock fueron ignorados hasta que a finales
de la década de 1960 varios laboratorios encontraron se forma casi simultánea
elementos móviles al interior del cromosoma circular de las bacterias. Fue en
este marco de referencia que los elementos transponibles de McClintock pasaron
a denominarse, transposones.
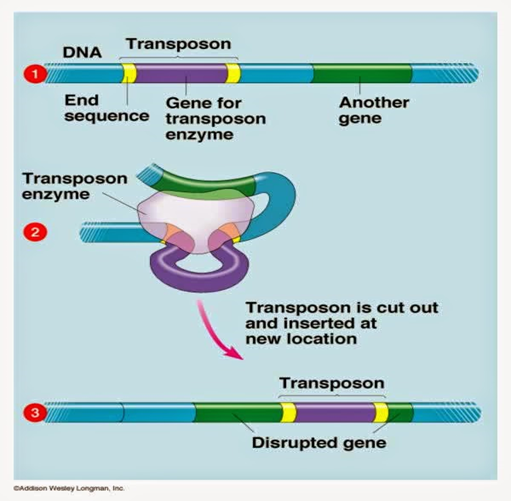
Un
transposón es un elemento que puede movilizarse al interior del genoma de los
seres vivos, y al contrario de lo que puede pensarse involucra la presencia de
varios
Figura
75. Enzima de transposición. Una de las características de cualquier mutación
es que es ciega, y la mayoría de las veces o causa enfermedades o es silenciada
por el genoma. Cuando un transposón "morado" se inserta en medio de
un gen "verde" puede causar enfermedades al destruir la función del
gen. (YouTube).
Esta
enzima puede literalmente catalizar el corte de todo el elemento móvil y
pegarlo en otra posición del genoma. Esto implica que los genes lejos de ser
elementos estructurales de un genoma, poseen una identidad propia, y pueden
incluso llegar a comportarse como pequeños individuos que aportan a un todo,
siendo capaces de moverse. Existe la posibilidad de que todo el elemento móvil
pueda duplicarse parcialmente gracias a la recombinación no paralela en la
meiosis I, lo cual genera nuevos transposones que a su vez pueden buscar otras
posiciones del genoma.
La
unidad a transponer aparte de codificar el locus de la transposasa y el locus
que va a movilizarse, debe estar flanqueada por una secuencia repetitiva que
marca la posición donde la transposasa debe pegarse. El primer paso es que la
transposasa reconozca las secuencias de flanqueo y se pegue, luego la corta
quedando una estructura con dos proteínas de transposasa en las puntas de una
secuencia de ADN cortado. La transposasa posee otro dominio que le permite
pegarse consigo misma, formando un elemento de ADN circular semejante a un
plásmido que se movilizará al interior del genoma.
Una vez encontrado un nuevo lugar, el complejo de
ADN-transposasa corta la hebra de ADN y media la inserción del elemento móvil.
El proceso también involucra una duplicación pequeña del ADN blanco que queda
flanqueando arriba y abajo al elemento móvil. Este tipo de duplicaciones sirve
para identificar los
Tal
como lo demostró originalmente McClintock, el genoma de los eucariotas posee
una cantidad relativamente alta de elementos móviles. De hecho, en el modelo
biológico humano, cerca del 45% del ADN al interior del núcleo puede asociarse
a elementos móviles. Esto implica que los elementos móviles tienden a perder su
función, e incluso la capacidad de movilizarse una vez que han experimentado el
cambio de lugar, lo cual va en concordancia con la naturaleza ciega de las
mutaciones tipo SNP.
Al
igual que ocurre con las duplicaciones del ADN total, cuando un transposón se
moviliza de un lugar a otro tiende a ser silenciado por el genoma mediante
cambios epigenéticos de las histonas o mediante metilaciones que bloquean su
expresión. Peor aún, muchos elementos móviles tienden a insertarse en medio de
Los
transposones al igual que cualquier gen deben venir de algún lugar, ¿cierto?
Existe otro tipo de elemento móvil que permite la duplicación parcial de
segmentos del ADN, a estos loci se los denomina retrotransposones.
Un retrotransposón a diferencia de un transposón, se mueve al interior del
genoma gracias a la maquinaria molecular normal, el loci completo se convertido a ARN mensajero “por lo que la copia
original queda intacta”, pero en esta posición, existe una proteína llamada ARN
transcriptasa reversa que transforma al ARN mensajero a ADN creando una copia
del gen, la cual procede a insertarse en otro lugar. Muchos retrovirus poseen
la misma enzima como los del VIH.
Familias
de genes como los de las globinas pueden ser explicadas como transposones y
retrotransposones. La retrotrasnposición da origen a la copia del gen, sin que
se requiera que todo el genoma se duplique. Posteriormente uno de los locus
puede transponerse a otro cromosoma, y allí volver a copiarse por
retrotransposición. Esto convierte a los genes que pueden copiarse y moverse en
entidades con cierta individualidad, de hecho, en las globinas dicha
individualidad puede verse en que todos esos
El
hecho de que muchos virus denominados retrovirus puedan realizar proezas
semejantes ha permitido generar hipótesis sobre la importancia de los virus en
la evolución biológica, ya que sin la enzima transcriptasa reversa, muchas
familias de genes no hubieran podido surgir. Lo anterior implica que al menos
los retrovirus al donar la enzima transcriptasa reversa han servido como parte
del verdadero motor evolutivo que es la variación aleatoria, ya que, al permitir una copia
de genes más fácil, aceleran el potencial para la creación de nuevos rasgos al
interior de los seres vivos.
Personalmente,
a diferencia de lo que proponen autores como Máximo Sandin, esto es
perfectamente concordante con el esquema de la síntesis evolutiva moderna,
después de todo los loci nuevos creados por la
retrotransposición deben medirse ante la selección natural cuando
subfuncionalizan o neofuncionalizan.
Las
secuencias moderadamente repetitivas pueden componer aproximadamente el 80% del
genoma de un eucariota y la mayoría de ellas pertenecen a familias de
pseudogenes relacionables a retrotransposones. De hecho, las dos familias de
secuencias moderadamente repetitivas más importantes denominadas Alu y L1 son
retrotransposones. Los L1 por ejemplo, poseen un locus que codifica para una
proteína con dos dominios, una transcriptasa reversa que transforma mensajeros
de ARN a ADN, y al mismo tiempo posee un dominio endonucleasa que permite a ese
ADN insertarse en el genoma. Lo interesante es que los humanos poseemos 500.000
copias de ese gen, aunque la mayoría de ellas se encuentra dañada debido a
mutaciones de tipo SNP. Ese valor de 500.000 copias contrasta con los 20.400 loci de genes que expresan proteínas.
Cuando
algo de este tamaño aparece en el genoma de un organismo, la primera pregunta
que viene a la mente s ¿cuál es su función? Muchos biólogos moleculares
pensaban que las secuencias moderadamente repetitivas eran principalmente
basura, de acuerdo con este punto de vista, los retrotransposones son una
especie simbionte comensalista a nivel molecular, que, a diferencia de un
virus, no mata, pero tampoco beneficia a su portador. Aunque este pudiera ser
su origen, o incluso asumiendo que los primeros retrotransposones fueran virus
degenerados, con un ciclo de infección reducido, no implica que no posean
función. El punto es que, aunque no posea una función para el individuo, los
retrotransposones si constituyen una fuente de variabilidad y potencial
evolutivo para linajes de seres vivos.
Los
elementos móviles pueden en ocasiones arrastrar partes del genoma del hospedero
moviéndolos de un lugar a otro. Este mecanismo cuando es muy limitado puede
permitir el traslado de dominios o módulos de un gen a otro gen, lo que genera
proteínas multimodulares nuevas. Cuando es más extenso puede duplicar uno o más
genes. Este proceso es facilitado en el genoma eucariota debido a los intrones,
si el segmento nuevo se inserta en un intrón, no se dañará la proteína generada
por el gen al cual se le ha adicionado el nuevo elemento. Muchas proteínas,
como las que están relacionadas con el flagelo bacteriano manifiestan evidencia
de haber surgido por este mecanismo.
Las
secuencias que emplean algunos elementos móviles para moverse de un lugar a
otro, han sido asociadas a los elementos reguladores del ADN. En otras
palabras, las secuencias que regulan la expresión de las proteínas en el genoma
eucariota tienen su origen en las secuencias que permiten la transposición de
los transposones. Adicional a lo anterior, muchos elementos móviles que carecen
de función poseen similitudes de secuencia con genes funcionales en otros
vertebrados, lo cual convierte al ADN basura en un registro fósil de nivel
molecular.
En
algunos casos, los elementos móviles poseen relaciones con genes activos y con
pseudogenes. La enzima telomerasa, la cual juega un papel preponderante en la
replicación final del ADN pudo haberse originado por retrotranposición de un
elemento móvil ancestral. La enzima que media la reorganización de las regiones
hipervariables de los anticuerpos en el sistema inmune también posee rastros de
ser un retrotransposon. La misma familia de las globinas muestra evidencias
simultáneas de transposición y retrotransposición.
Un
punto es claro, la retrontransposición y la transposición poseen un efecto
profundo en el genoma de los eucariotas y procariotas. Resulta interesante ver
que unas décadas atrás la mayoría de los biólogos moleculares concebía al
genoma como una entidad muy fija y de difícil cambio, lo cual complicaba
algunos aspectos de la teoría de la evolución.
De
hecho, la propuesta original de la TSE se basaba en que el genoma solo podía
experimentar cambios muy leves de una generación a otra, pues de lo contrario,
su estabilidad general se vería muy afectada. Esto a su vez concluyó en la
propuesta del gradualismo filético a ultranza, o lo que es lo mismo, concebir a
la evolución como un proceso absolutamente gradual. Estos descubrimientos han demostrado que el
genoma posee una flexibilidad y resiliencia muy grandes, lo cual facilita
explicaciones evolutivas, en otras palabras, el potencial evolutivo de los
seres vivos es mayor de lo que se habría esperado originalmente. Gracias a
estos descubrimientos en 1983 se le otorgó a Barbara McClintock el Premio
Nobel.
La
respuesta a la pregunta ¿de dónde surgen los genes? Parecía muy clara cuando
comenzó en nuevo milenio, y la respuesta era, por duplicación de genes previos.
Esto es porque aun el biólogo que confiaba más en el azar vería muy improbable
que los genes pudiesen evolucionar por SNP de secuencias no codificantes sin
regiones promotoras o reguladoras.
Sin
embargo en 2006 algo sucedió que estremeció los fundamentos de la biología
molecular realizado por Yuuki Hayashi,
Takuyo Aita, Hitoshi Toyota, Yuzuru Husimi, Itaru Urabe y Tetsuya Yomo (Hayashi et al., 2006) en
el cual demuestran experimentalmente el potencial de un gen de emerger del caos
del ADN no codificante. Debido a la imposibilidad teórica, el hecho de que
suceda en la práctica demuestra como los procesos de variación aleatoria SNP y
la selección natural son capaces de crear información prácticamente de la nada.
A este
tipo de genes que emergen de mutaciones aleatorias sin un gen previo que sirva
de base se los denomina genes de Novo y desde entonces han sido reportados
incluso en el genoma humano.
Los
genes nuevos son las principales fuentes de innovación en los genomas. Sin
embargo, hasta años recientes el entendimiento de como los genes nuevos se
originaban y como evolucionaban una vez habían emergido brillaba por la
ausencia de apropiadas bases de datos moleculares “y muchos lo siguen pensando
así”. Con el advenimiento de la era genómica llegó una revolución en la
información disponible para estudiar genes nuevos. Por primera vez, décadas de
principios teóricos pueden ser medidos de manera empírica, junto con
nuevas líneas de investigación Aunque la totalidad del artículo en que se
basa la anterior introducción será sujeto de un análisis futuro, en este punto
me enfocaré únicamente en tres tópicos de manera muy muy resumida, la
duplicación de genes, la subfuncionalización y más importante aún, la
neofuncionalización (Cardoso-Moreira & Long, 2012). Aunque existen otras formas para la aparición de genes
nuevos, el asunto empírico es que muchos de los genes de los sistemas complejos
de múltiples componentes integrados que interactúan entre si poseen líneas de
evidencia que los implica como genes duplicados que han experimentado
subfuncionalización y neofuncionalizacion. Es importante recalcar que, si bien
la primera parte de la explicación para los sistemas complejos es la
duplicación, la actual estrategia del DI como bien lo expone Behe (William & Ruse, 2004) es básicamente negar la otra mitad de la historia, la
neofuncionalización y la subfuncionalización.
Durante
la duplicación de genes, tenemos una secuencia de ADN que al pasar por una
mutación se duplica en una misma célula y luego esa duplicación es mantenida en
el resto de los descendientes. En tal caso, hay que distinguir entre dos tipos
diferentes, la duplicación a gran escala de genomas completos denominada
poliploidia de la duplicación de genes o secuencias de ADN más pequeñas (Conant & Wolfe, 2008). Un detalle a rescatar es que, la
historia subsecuente de un gen duplicado “la cual parecen querer olvidar, los
defensores del DI y del creacionismo” es que dependerá de qué tipo de evento
duplicador la hubiera generado, es decir, los genes que se fijen y su historia
diferirá de si nacieron como consecuencia de una duplicación a pequeña escala o
por un evento de poliploidia (Conant & Wolfe, 2008; Maere et al.,
2005; Wapinski, Pfeffer, Friedman, & Regev, 2007). Los genes duplicados representan una porción bastante
grande de los genomas de diferentes tipos de eucariontes, por ejemplo, el 17%
en algunas bacterias, al 65% en la planta Arabidopsis (Zhang,
2003). La diversidad de las
duplicaciones es amplia y no se restringe a los polos opuestos de, un solo gen
o el genoma completo, y de hecho es bastante común que se den duplicaciones de
grupos de genes (Cardoso-Moreira & Long, 2012).
Un detalle interesante
sobre la determinación de la duplicación de genes, resulta de cumplir a
cabalidad las hipótesis originales de Darwin. A medida que los duplicados son
más viejos y han tenido más tiempo para mutar, estos se hacen más diferentes,
mientras que los duplicados más jóvenes son más semejantes entre sí. El
problema de tener genes duplicados muy semejantes, es que los métodos
moleculares actuales los hacen colapsar cuando se ensamblan los genomas
completos, y por lo tanto los hacen pasar como si fueran un solo gen. Como
resultado, el número de genes duplicados recientemente es mucho más alto de lo
que puede llegar a medirse (Bailey et al., 2002). No
nos enfocaremos en los mecanismos moleculares de las duplicaciones ya que los
proponentes de DI parecen no cuestionar la existencia de la duplicación.
Ahora, digamos que, en
lugar de tener un gen con una función específica, afinada, especifica y
completamente irrenunciable llamada A, después del evento duplicador tenemos
dos genes que expresan la misma condenada proteína. ¿Que pasara? Aparentemente según los escritores del diseño
inteligente, no mucho, ya que, para ellos, un gen duplicado es solo información
redundante. Una vez se ha duplicado un gen ocurre el fenómeno clásico
denominado compensación de dosis. Y digo clásico porque su efecto es demostrable
aun desde los primeros trabajos en genética, con la genética clásica
mendeliana. La compensación de dosis se presenta cuando se tienen dos
Los genes duplicadores
experimentan la misma cuestión, en el momento en que los genes se duplican, uno
compensará la necesidad del otro, por lo que, aun cuando la función A sea
completamente irrenunciable, esta podrá llevarse a cabo por la presencia de uno
de los dos loci, mientras que el otro podrá acumular mutaciones. La
acumulación de mutaciones conllevará a tres destinos posibles, “la
neutralización perdida de función o pseudogenización; la subfuncionalización y
la neofuncionalización” (Cardoso-Moreira & Long, 2012). Dada la condenada aleatoriedad de las mutaciones, la ruta más
común para el gen duplicado es la pseudogenización, lo que implica la perdida
de la función del gen, lo que lo hace ingresar en la categoría de genes no
codificantes, o bajo el infortunado nombre de ADN basura. El asunto puede ser
tan severo, que se ha estimado que existe al menos un pseudo gen por cada ocho
genes en el gusano C. elegans (Harrison, Echols, &
Gerstein, 2001), y
para los humanos, “cuya cascada de coagulación intentamos explicar” la cantidad
de pseudogenes es de uno por cada dos genes funcionales (Harrison et al., 2001).
Es importante resaltar
que, no todos los pseudogenes se generan por duplicaciones, muchos pseudogenes
han aparecido por la pérdida de presiones de selección, por ejemplo, las
pérdidas de la sensibilidad del olfato en muchos primates han dado origen a la
pseudogenizacion de los genes responsables por un buen olfato (Rouquier, Blancher, & Giorgi, 2000). El
asunto es que teóricamente, el resultado más probable de la vasta mayoría de
mutaciones es el de mutaciones deletéreas, o neutrales. Pero aun así… Las
duplicaciones pueden generar neofuncionalizaciones, e modelo fue ya propuesto
por Ohno bajo el argumento de la compensación de dosis (Ohno, 1970). Lo
interesante de la selección natural es que actúa como un filtro amplificador o
un trinquete irreversible; una vez que ocurre una rara mutación ventajosa, esta
es fijada y amplificada rápidamente en el interior de la población (Cardoso-Moreira & Long, 2012). Un ejemplo, ahora clásico de nefuncionalización ocurrió en
una especie de monos vegetarianos, en la cual se duplicó el gen de la
ribonucleasa. Después de la duplicación, el gen duplicado sufrió una serie de
mutaciones que conllevaron a generar una función digestiva más efectiva en un
nuevo microambiente (Zhang, Zhang, & Rosenberg, 2002)
Más DULCE aun, recientes
datos genéticos sugieren que las nuevas funciones pueden darse de manera más
común, y aún más, que pueden emerger al mismo tiempo en que nace el gen
duplicado. De hecho, resulta interesante que el único tipo de mutaciones
duplicantes que dejan intactas a los genes es la poliploidia, los demás
mecanismos de duplicación alteran de alguna forma a los genes durante el
proceso “duplicaciones parciales, fisiones de genes, cambio de marco de lectura
de exones, retrotrasposones, y la más rara de todas las rarezas teóricas genes
DE NOVO” (Cardoso-Moreira & Long, 2012). Finalmente hay que tener en cuenta a la epigenética; aun
cuando la secuencia genética del
Bueno ya esto se
extendió mucho y tratar el tema completo requeriría su propia serie de
artículos, así que resumiré brevemente la subfubncionalización. En ocasiones la
proteína original lleva a cabo dos funciones de manera rudimentaria, la
subfuncionalización le permite a los





No hay comentarios:
Publicar un comentario