(Ciencias de Joseleg) (Biología) (Teoría de la Biología) (Genética moderna) (Introducción) (Naturaleza química del gen) (Conflicto por la estructura del ADN) (Estructura del ADN) (La replicación del ADN) (Síntesis de proteínas) (Denaturación y renaturación) (Estructura del genoma) (Tipos de mutaciones) (Elementos móviles del genoma) (Identificación humana) (Referencias bibliográficas)
En muchos sentidos, el progreso en la biología durante el siglo pasado se refleja en nuestro concepto cambiante del gen. Como resultado del trabajo de Mendel, los biólogos aprendieron que los genes son elementos discretos que rigen la aparición de rasgos específicos. Los mendelianos defendieron el concepto de que un gen determina un rasgo. Boveri, Weismann, Sutton y sus contemporáneos descubrieron que los genes tienen una encarnación física como partes del cromosoma. Morgan, Sturtevant y sus colegas demostraron que los genes tienen ubicaciones específicas: residen en ubicaciones particulares en cromosomas particulares, y estas ubicaciones permanecen constantes de un individuo de una especie a la siguiente. Griffith, Avery, Hershey y Chase demostraron que los genes estaban compuestos de ADN, y Watson y Crick resolvieron el rompecabezas de la estructura del ADN, que explicaba cómo esta notable macromolécula podría codificar información hereditaria. Aunque las formulaciones de estos conceptos fueron hitos en el camino hacia la comprensión genética, ninguno de ellos abordó el mecanismo por el cual la información almacenada en un gen se pone a funcionar para gobernar las actividades celulares. Este es el tema principal que se discutirá en el presente capítulo. Comenzaremos con conocimientos adicionales sobre la naturaleza de un gen, lo que nos acerca a su papel en la expresión de rasgos heredados.
Figura 46. De
izquierda a derecha, primera fila: Archibald Garrod, George Beadle; segunda
fila: Edward Tatum y François Jacob.
La
primera percepción significativa sobre la función genética fue obtenida por
Archibald Garrod 1857-1936, un médico escocés que informó en 1908 que los
síntomas exhibidos por personas con ciertas enfermedades hereditarias raras
fueron causados por la ausencia de enzimas específicas. Una de las
enfermedades investigadas por Garrod fue la alcaptonuria, una afección que se
diagnostica fácilmente porque la orina se oscurece al exponerse al aire. Garrod
descubrió que las personas con alcaptonuria carecían de una enzima en su sangre
que oxidaba el ácido homogentísico, un compuesto formado durante la
descomposición de los aminoácidos fenilalanina y tirosina. A medida que se
acumula el ácido homogentísico, se excreta en la orina y se oscurece en color
cuando se oxida por el aire. Garrod había descubierto la relación entre un
defecto genético, una enzima específica y una condición metabólica específica.
Llamó a tales enfermedades "errores innatos del metabolismo". Como
parece haber sucedido con otras observaciones tempranas de importancia básica
en genética, los hallazgos de Garrod no fueron apreciados durante décadas.
La idea
de que los genes dirigen la producción de enzimas fue resucitada en la década
de 1940 por George Beadle 1903-1989 y Edward Tatum 1909-1975 del Instituto de
Tecnología de California. Estudiaron Neurospora, un moho de pan tropical
que crece en un medio muy simple que contiene una sola fuente de carbono
orgánico (por ejemplo, un azúcar), sales inorgánicas y biotina (vitamina B).
Debido a que necesita tan poco para vivir, se supuso que Neurospora
sintetiza todos sus metabolitos. Beadle y Tatum razonaron que un organismo con
una capacidad sintética tan amplia debería ser muy sensible a las deficiencias
enzimáticas, que deberían detectarse fácilmente utilizando el protocolo
experimental adecuado.
El plan
de Beadle y Tatum era irradiar esporas de moho y detectar mutaciones que
causaran que las células carecieran de una enzima particular. Para detectar
estas mutaciones, se probaron las esporas irradiadas para determinar su
capacidad de crecer en un medio mínimo que carecía de los compuestos esenciales
conocidos por ser sintetizados por este organismo. Si una espora no puede
crecer en un medio mínimo pero una espora genéticamente idéntica puede crecer
en un medio suplementado con una coenzima particular (p. Ej., Ácido pantoténico
de la coenzima A), los investigadores podrían concluir que las células tienen
una deficiencia enzimática que les impide de sintetizar este compuesto
esencial.
Beadle y Tatum comenzaron irradiando más de mil células. Dos de las esporas demostraron ser incapaces de crecer en el medio mínimo: una necesitaba piridoxina (vitamina B6) y la otra necesitaba tiamina (vitamina B1). Eventualmente, se probó la progenie de aproximadamente 100.000 esporas irradiadas, y se aislaron docenas de mutantes. Cada mutante tenía un defecto genético que producía una deficiencia enzimática que impedía que las células catalizaran una reacción metabólica particular. Los resultados fueron claros: un gen lleva la información para la construcción de una enzima particular. Esta conclusión se conoció como la hipótesis de "un gen-una enzima". Una vez que se supo que las enzimas a menudo están compuestas de más de un polipéptido, cada uno de los cuales está codificado por su propio gen, el concepto se modificó a "un polipéptido un gen". Aunque esta relación sigue siendo una aproximación cercana de la función básica de un gen, también ha tenido que ser modificado debido al descubrimiento de que un solo gen a menudo genera una variedad de polipéptidos, principalmente como resultado de un empalme “splicing” alternativo. También sería evidente que la descripción de un gen estrictamente como un almacén de información para polipéptidos es una definición demasiado estricta. Muchos genes codifican moléculas de ARN que, en lugar de contener información para la síntesis de polipéptidos, funcionan como ARN en sí mismas. Con esto en mente, podría ser mejor definir un gen como un segmento de ADN que contiene la información para una cadena polipeptídica única o para uno o más ARN funcionales.
Figura 47. De
izquierda a derecha: Jacques Monod, Sydney Brenner y Matthew Meselson.
La
información presente en un segmento de ADN se pone a disposición de la célula
mediante la formación de una molécula de ARN. La síntesis de un ARN a partir de
una plantilla de ADN se denomina transcripción. El término transcripción
denota un proceso en el que la información codificada en las cuatro letras de
desoxirribonucleótidos de ADN se reescribe, o se transcribe, en un lenguaje
similar compuesto por cuatro letras de ribonucleósidos de ARN. Examinaremos el
mecanismo de la transcripción en breve, pero primero continuaremos con el papel
de un gen en la formación de polipéptidos.
Los estudios llevados a cabo en la década de 1950 descubrieron la relación entre la información genética y la secuencia de aminoácidos, pero este conocimiento en sí mismo no proporcionaba ninguna pista sobre el mecanismo por el cual se genera la cadena polipeptídica específica. Como ahora sabemos, hay un intermedio entre un gen y su polipéptido; el intermediario es el ARN mensajero (ARNm). El descubrimiento trascendental del ARNm fue realizado en 1961 por François Jacob 1920-2013 y Jacques Monod 1910-1976 del Instituto Pasteur en París, Sydney Brenner 1927-Vivo de la Universidad de Cambridge y Matthew Meselson 1930-vivo del Instituto de Tecnología de California. Un ARN mensajero se ensambla como una copia complementaria de una de las dos cadenas de ADN que componen un gen. Debido a que su secuencia de nucleótidos es complementaria a la del gen del cual se transcribe, el ARNm conserva la misma información para el ensamblaje de polipéptidos que el gen mismo. Por esta razón, un ARNm también se puede describir como una cadena de "sentido" o un ARN de codificación. Los ARNm eucariotas no se sintetizan en su forma final (o madura) sino que, en su lugar, se deben tallar (o procesar) a partir de pre-ARNm mucho más grandes.
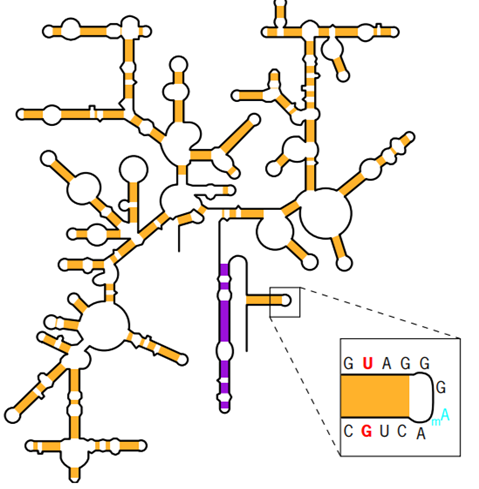
Figura
48. Estructura bidimensional de un ARN
ribosómico bacteriano. Muestra el
emparejamiento de bases extensivo entre las diferentes regiones de la cadena
simple. La sección expandida muestra la secuencia de bases de un tallo y un
bucle, que incluye un par de bases no estándar (G-U) y un nucleótido
modificado, metiladenosina. Una de las hélices está sombreada de forma
diferente porque juega un papel importante en la función de los ribosomas.
El uso
de ARN mensajero permite a la célula separar el almacenamiento de información
de la utilización de la información. Mientras que el gen permanece almacenado
en el núcleo como parte de una enorme molécula de ADN estacionaria, su
información se puede transmitir a un ácido nucleico móvil mucho más pequeño que
pasa al citoplasma. Una vez en el citoplasma, el ARNm puede servir como
plantilla para dirigir la incorporación de aminoácidos en un orden particular
codificado por la secuencia de nucleótidos del ADN y ARNm. El uso de un ARN
mensajero también permite que una célula amplifique en gran medida la emisión
de polipéptidos. Una molécula de ADN puede servir como plantilla en la
formación de muchas moléculas de ARNm, cada una de las cuales puede usarse en
la formación de un gran número de cadenas de polipéptidos.
Las
proteínas se sintetizan en el citoplasma mediante un complejo proceso llamado traducción. La traducción requiere la
participación de docenas de diferentes componentes, incluidos los ribosomas.
Los ribosomas son "máquinas" citoplásmicas complejas que se pueden
programar, como una computadora, para traducir la información codificada por
cualquier ARNm. Los ribosomas contienen proteínas y ARN. Los ARN de un ribosoma
se llaman ARN ribosómicos (ARNr) y, al igual que los ARNm, cada uno se
transcribe a partir de una de las cadenas de ADN de un gen. En lugar de
funcionar como una entidad informática i digital, los rRNA proporcionan soporte
estructural analógico y catalizan la reacción química en la que los aminoácidos
se unen covalentemente entre sí. Los ARN de transferencia (o ARNt) constituyen
una tercera clase importante de ARN que se requiere durante la síntesis de
proteínas. Se requieren ARN de transferencia para traducir la información en el
código de nucleótidos de ARNm en el "alfabeto" de aminoácidos de un
polipéptido.
Tanto los rRNA como los tRNA deben su actividad a sus
complejas estructuras secundarias y terciarias. A diferencia del ADN, que tiene
una estructura helicoidal bicatenaria similar, independientemente de la fuente,
muchos ARN se pliegan en una forma tridimensional compleja, que es marcadamente
diferente de un tipo de ARN a otro. Por lo tanto, al igual que las proteínas,
los ARN llevan a cabo una amplia gama de funciones debido a sus diferentes
formas. Al igual que con las proteínas, el plegamiento de moléculas de ARN
sigue ciertas reglas. Mientras que el plegamiento de la proteína es impulsado
por la extracción de residuos hidrófobos en el interior, el plegamiento de ARN
es impulsado por la formación de regiones que tienen pares de bases
complementarias. Las regiones con pares de bases típicamente forman
"vástagos" bicatenarios (y de doble hélice), que están conectados a
"bucles" monocatenarios. A diferencia del ADN, que consiste
exclusivamente en Watson-Crick estándar (GC, AT) pares de bases, los ARN a
menudo contienen pares de bases no estándar y bases nitrogenadas modificadas.
Estas regiones no ortodoxas de la molécula sirven como sitios de reconocimiento
de proteínas y otros ARN, promueven el plegamiento de ARN y ayudan a
estabilizar la estructura de la molécula. La importancia del apareamiento de
bases complementario se extiende mucho más allá de la estructura de tRNA y
rRNA. Como veremos a lo largo de este capítulo, el emparejamiento de bases
entre las moléculas de ARN desempeña un papel central en la mayoría de las
actividades en las que los ARN están comprometidos.
Los roles de mRNAs, rRNAs y tRNAs se exploran en detalle
en las siguientes secciones de este capítulo. Las células eucariotas forman una
serie de otros ARN, que también desempeñan papeles vitales en el metabolismo
celular; estos incluyen ARN nucleares pequeños (snARN), ARN nucleolares
pequeños (ARNs sno), ARN pequeños de interferencia (ARNip), ARNip, microARN
(miARN) y una variedad de otros ARN no codificantes, es decir, ARN que no
contienen información para aminoácidos. La mayoría de estos ARN cumple una
función reguladora, controlando varios aspectos de la expresión génica, o
incluso sirviendo como defensa contra infecciones virales. Varios de estos ARN
han irrumpido en la escena en los últimos años, recordándonos una vez más que
hay muchas actividades celulares ocultas esperando a ser descubiertas.
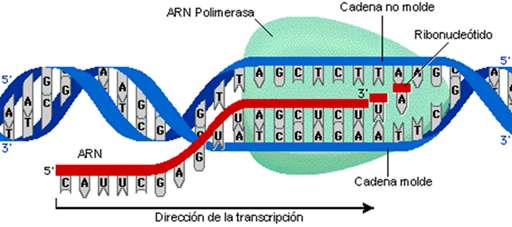
La transcripción es un proceso en el que una cadena de ADN proporciona la información para la síntesis de una cadena de ARN mensajero. Las enzimas responsables de la transcripción tanto en células procariotas como eucariotas se denominan ARN polimerasas dependientes de ADN, o simplemente ARN polimerasas. Estas enzimas son capaces de incorporar nucleótidos, uno a la vez, en una cadena de ARN cuya secuencia es complementaria a una de las cadenas de ADN, que sirve como plantilla.
Figura
49. Resumen de la
transcripción del ARN (YouTube).
El
primer paso en la síntesis de un ARN es la asociación de la polimerasa con la
plantilla de ADN. Esto trae a colación una cuestión de interés más general, a
saber, las interacciones específicas de dos macromoléculas, proteínas y ácidos
nucleicos, químicamente muy diferentes. Del mismo modo que diferentes proteínas
han evolucionado para unir diferentes tipos de sustratos y catalizar diferentes
tipos de reacciones, también han evolucionado algunos de ellos para reconocer y
unirse a secuencias específicas de nucleótidos en una cadena de ácido nucleico.
El sitio en el ADN al que se une una molécula de ARN polimerasa antes de
iniciar la transcripción se denomina promotor. Las ARN polimerasas
celulares no son capaces de reconocer los promotores por sí mismas, pero
requieren la ayuda de proteínas adicionales llamadas factores de
transcripción.
Además de proporcionar un sitio de unión para la polimerasa, el promotor
contiene la información que determina cuál de las dos cadenas de ADN se
transcribe y el sitio en el que comienza la transcripción. La ARN polimerasa se
mueve a lo largo de la cadena de ADN molde hacia su extremo 5 (es decir, en una
dirección de 3 a 5). A medida que la polimerasa progresa, el ADN se desenrolla
temporalmente y la polimerasa ensambla una cadena complementaria de ARN que
crece desde su extremo 5 en una dirección 3.
La ARN polimerasa cataliza la reacción altamente
favorable en la que los sustratos de ribonucleósidos trifosfato (NTP) se
escinden en nucleósidos monofosfatos a medida que se polimerizan en una cadena
covalente. Las reacciones que conducen a la síntesis de ácidos nucleicos (y
proteínas) son intrínsecamente diferentes de las del metabolismo intermediario.
Considerando que algunas de las reacciones que conducen a la formación de moléculas
pequeñas, como aminoácidos, pueden estar lo suficientemente cerca del
equilibrio se puede medir una reacción inversa considerable, aquellas
reacciones que conducen a la síntesis de ácidos nucleicos y proteínas deben
ocurrir en condiciones en las que prácticamente no hay reacción inversa. Esta
condición se cumple durante la transcripción con la ayuda de una segunda
reacción favorable catalizada por una enzima diferente, una pirofosfatasa. En
este caso, el pirofosfato (PPi) producido en la primera reacción se hidroliza a
fosfato inorgánico (Pi). La hidrólisis del pirofosfato libera una gran cantidad
de energía libre y hace que la reacción de incorporación de nucleótidos sea
esencialmente irreversible.
A medida que la polimerasa se mueve a lo largo de la plantilla
de ADN, incorpora nucleótidos complementarios en la cadena de ARN en
crecimiento. Se incorpora un nucleótido en la cadena de ARN si es capaz de
formar un par de bases adecuado (Watson-Crick) con el nucleótido en la cadena
de ADN que se transcribe, solo cambia la timina por uracilo, pero sigue las
mismas reglas de Chargaff. Una vez que
la polimerasa ha pasado un determinado tramo de ADN, la doble hélice del ADN se
re-forma a su condición de no transcripción. En consecuencia, la cadena de ARN
no permanece asociada con su plantilla como un híbrido de ADN-ARN (excepto por
aproximadamente nueve nucleótidos justo detrás del sitio donde opera la
polimerasa). Las ARN polimerasas son capaces de incorporar de aproximadamente
20 a 50 nucleótidos en una molécula de ARN en crecimiento por segundo, y muchos
genes en una célula se transcriben simultáneamente por cien o más polimerasas.
La frecuencia con la que se transcribe un gen está estrechamente regulada y
puede variar drásticamente dependiendo del gen dado y las condiciones
prevalecientes.
Figura
50. Editando el mensaje. Las proteínas funcionan como bloques ensamblables
llamados dominios, los dominios pueden estar separados por secuencias que no
van en la proteína final y deben ser eliminados por el empalme diferencial del
ARN mensajero (YouTube). Se ha descubierto que un mensajero puede
editarse alternativamente a varios mensajes maduros diferentes.
Mientras que la
transcripción de genes codificadores de proteínas procariotas crea ARN
mensajero (ARNm) que está listo para la traducción a proteínas, la
transcripción de genes eucarióticos deja una transcripción primaria de ARN
(pre-ARNm), que primero tiene que someterse a una serie de modificaciones para
convertirse en un ARNm maduro.
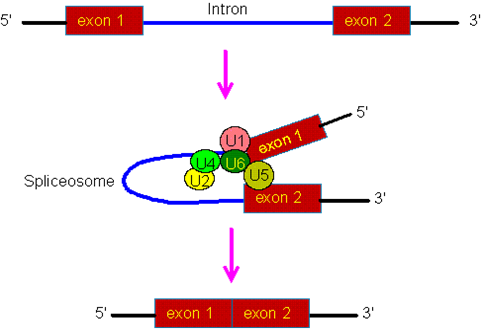
Una
modificación muy importante del pre-ARNm eucariótico es el empalme de ARN. La
mayoría de los pre-ARNm eucariotas consisten en segmentos alternantes llamados
exones e intrones. Durante el proceso de empalme, un complejo catalítico de
ARN-proteína conocido como spliceosome cataliza dos reacciones de
transesterificación, que eliminan un intrón y lo liberan en forma de estructura
lariat, y luego unen los exones adyacentes.
Un intrón es cualquier secuencia de nucleótidos dentro de
un gen que se elimina mediante corte y empalme de ARN durante la maduración del
producto de ARN final. El término intrón se refiere tanto a la secuencia de ADN
dentro de un gen como a la secuencia correspondiente en las transcripciones de
ARN. Las secuencias que se unen en el ARN maduro final después del corte y
empalme del ARN son exones. Los intrones se encuentran en los genes de la
mayoría de los organismos y muchos virus, y pueden ubicarse en una amplia gama
de genes, incluidos los que generan proteínas, en el ARN ribosómico (ARNr) y en
el ARN de transferencia (ARNt). Cuando las proteínas se generan a partir de
genes que contienen intrones, el empalme de ARN tiene lugar como parte de la
ruta de procesamiento del ARN que sigue a la transcripción y precede a la
traducción. La palabra intrón se deriva del término región intragénica, es
decir, una región dentro de un gen.
En ciertos casos, algunos intrones o exones pueden eliminarse o retenerse en ARNm maduro. Este llamado splicing alternativo y crea series de transcritos diferentes que se originan a partir de un único gen.
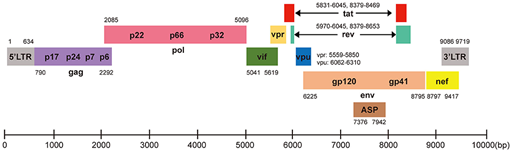
Figura
51. Sobrelapamiento de loci en un genoma. Diagrama de cajas/dominios de las proteínas del
VIH, siendo estas: gag, pol, vif, vpr, vpu, tat, rev, env, y nef. Como se puede
observar varios loci
Debido a que estas transcripciones se pueden traducir
potencialmente a proteínas diferentes, el corte y empalme extiende la
complejidad de la expresión del gen eucariótico. El procesamiento extenso del
ARN puede ser una ventaja evolutiva hecha posible por el núcleo de eucariotas.
En procariotas, la transcripción y la traducción suceden juntas, mientras que,
en los eucariotas, la membrana nuclear separa los dos procesos, dando tiempo
para que ocurra el procesamiento del ARN.
El empalme alternativo trae como consecuencia que un solo
gen pueda almacenar información para más de una unidad funcional sea esta ARN o
un polipéptido, incluso puede darse el caso de que dos loci almacenen
entre los dos la información de un tercer loci fantasma
como ocurre en el genoma de virus de ARN como el VIH causante del SIDA.
En eucariotas, el ARN mensajero maduro posterior a los
empalmes debe exportarse al citoplasma desde el núcleo. Mientras que algunos
ARN funcionan en el núcleo, muchos ARN se transportan a través de los poros
nucleares hacia el citosol. En particular, esto incluye todos los tipos de ARN
implicados en la síntesis de proteínas. En algunos casos, los ARN se
transportan adicionalmente a una parte específica del citoplasma, como una
sinapsis; luego son remolcados por proteínas motoras que se unen a través de proteínas
enlazadoras a secuencias específicas (llamadas "códigos postales") en
el ARN.
Figura 52. George Gamow y Marshall Nirenberg.
Para algunos ARN (ARN no codificante), el ARN maduro es
el producto génico final. En el caso del ARN mensajero (ARNm), el ARN es un
portador de información que codifica la síntesis de una o más proteínas de
manera analógica. El ARNm que porta una única secuencia de proteína (común en
eucariotas) es monocistrónico, mientras que el ARNm que porta múltiples
secuencias de proteínas (común en procariotas) se conoce como policistrónico.
Cada ARNm consta de tres partes: una región 5 'no
traducida (5'UTR), una región de codificación de proteínas o un marco de
lectura abierto (ORF), y una región 3' no traducida (3'UTR). La región de
codificación contiene información para la síntesis de proteínas codificada por
el código genético para formar tripletes. Cada triplete de nucleótidos de la
región codificante se denomina codón y corresponde a un sitio de unión
complementario a un triplete anticodón en ARN de transferencia. Los ARN de
transferencia con la misma secuencia de anticodón llevan siempre un tipo
idéntico de aminoácido. Los aminoácidos se encadenan juntos por el ribosoma de
acuerdo con el orden de tripletes en la región codificante. El ribosoma ayuda a
transferir ARN para unirse al ARN mensajero y toma el aminoácido de cada ARN de
transferencia y produce una proteína sin estructura. Cada molécula de ARNm se
traduce en muchas moléculas de proteína, en promedio ~ 2800 en mamíferos.
Una vez que se
reveló la estructura del ADN en 1953, se hizo evidente que la secuencia de
aminoácidos en un polipéptido estaba especificada por la secuencia de
nucleótidos en el ADN de un gen. Parecía muy poco probable que el ADN pudiera
servir como una plantilla física directa para el ensamblaje de una proteína. En
cambio, se supuso que la información almacenada en la secuencia de nucleótidos
estaba presente en algún tipo de código genético. Con el descubrimiento del ARN
mensajero como un intermediario en el flujo de información desde el ADN a la
proteína, la atención se centró en la forma en que una secuencia escrita en un
"alfabeto" de ribonucleótidos podría codificar una secuencia en un
"alfabeto" que consta de aminoácidos.
Uno de
los primeros modelos del código genético fue presentado por el físico George
Gamow 1904-1968, quien propuso que cada aminoácido en un polipéptido estaba
codificado por tres nucleótidos secuenciales. En otras palabras, las palabras
de código, o codones, para aminoácidos eran tripletas de nucleótidos. Gamow
llegó a esta conclusión con un poco de lógica de deductiva matemática. Él
razonó que requeriría al menos tres nucleótidos para que cada aminoácido
tuviera su propio codón único. Considere la cantidad de palabras que pueden
escribirse usando un alfabeto que contiene cuatro letras diferentes que
corresponden a las cuatro posibles bases que pueden estar presentes en un sitio
particular en el ADN (o ARNm). Hay 4 palabras posibles de una letra 4=41;
16=42 palabras posibles de dos letras; y 64 =43 palabras
posibles de tres letras. Debido a que hay 20 aminoácidos diferentes (palabras)
que deben especificarse, los codones deben contener al menos 3 nucleótidos
sucesivos (letras), evidentemente quedan varias sobrantes para sinónimos. La
naturaleza de triplete del código fue pronto verificada en una serie de
experimentos genéticos perspicaces conducidos por Francis Crick, Sydney Brenner
y sus colegas de la Universidad de Cambridge.
En 1961, se conocían las propiedades generales del
código, pero no se había descubierto una de las asignaciones de codificación de
los tripletes específicos. En ese momento, la mayoría de los genetistas
pensaban que tomaría muchos años descifrar todo el código. Pero Marshall
Nirenberg 1927-2010 y Heinrich Matthaei 1929-vivo lograron un avance que
utilizó una enzima polinucleótido fosforilasa para sintetizar
|
Figura 53. Código
genético. Gráfico del decodificador universal para el código
genético. El código es el mismo para todos los seres vivos, y le permite a
los virus atacar a sus anfitriones. |
El examen del diagrama
de codones en la Figura 67
indica que las asignaciones de aminoácidos son claramente no aleatorias. Si uno
busca las cajas de codones para un aminoácido específico, tienden a agruparse
dentro de una porción particular de la tabla. La agrupación refleja la
similitud en los codones que especifican el mismo aminoácido. Como resultado de
esta similitud en la secuencia del codón, las mutaciones espontáneas que causan
cambios de bases únicos en un gen a menudo no producirán un cambio en la
secuencia de aminoácidos de la proteína correspondiente. Se dice que un cambio
en la secuencia de nucleótidos que no afecta a la secuencia de aminoácidos es
sinónimo, mientras que un cambio que causa una sustitución de aminoácidos se
dice que es no sinónimo. Estos dos tipos de mutaciones describe varios de los
diferentes tipos de mutaciones discutidas en este capítulo. Los cambios
sinónimos tienen muchas menos probabilidades de modificar el fenotipo de un
organismo que los cambios no anónimos. En consecuencia, es mucho más probable
que los cambios no sinónimos sean seleccionados a favor o en contra por
selección natural. Ahora que hemos secuenciado los genomas de organismos
relacionados, como los de chimpancés y humanos, podemos observar directamente
las secuencias de genes homólogos y ver cuántos cambios son sinónimos o no
sinónimos. Es probable que los genes que poseen un exceso de sustituciones no
sinónimas en sus regiones codificadoras hayan sido influidos por la selección
natural.
El aspecto de
"salvaguardia" del código va más allá de su degeneración. Las
asignaciones de codones son tales que los aminoácidos similares tienden a ser
especificados por codones similares. Por ejemplo, los codones de los diversos
aminoácidos hidrofóbicos se agrupan en las dos primeras columnas del cuadro. En
consecuencia, una mutación que da como resultado una sustitución de bases en
uno de estos codones es probable que sustituya un residuo hidrófobo por otro.
Además, las mayores similitudes entre los codones relacionados con aminoácidos
se producen en los dos primeros nucleótidos del triplete, mientras que la mayor
variabilidad se produce en el tercer nucleótido. Por ejemplo, la glicina está
codificada por cuatro codones, todos los cuales comienzan con los nucleótidos
GG. Una explicación para este fenómeno se revela en la siguiente sección en la
que se describe el papel de los ARN de transferencia.
Los ácidos nucleicos y las proteínas son como dos idiomas
escritos con diferentes tipos de letras. Esta es la razón por la cual la
síntesis de proteínas se denomina traducción. La traducción requiere que la
información codificada en la secuencia de nucleótidos de un ARNm se decodifique
y se use para dirigir el ensamblaje secuencial de aminoácidos en una cadena
polipeptídica. La decodificación de la información en un ARNm se lleva a cabo
mediante ARN de transferencia, que actúan como adaptadores. Por un lado, cada
ARNt está unido a un aminoácido específico (como un ARNt aa), mientras que, por
otro lado, ese mismo ARNt es capaz de reconocer un codón particular en el ARNm.
La interacción entre codones sucesivos en el ARNm y aa-ARNt específicos conduce
a la síntesis de un polipéptido con una secuencia ordenada de aminoácidos.
Los ARN de transferencia
traducen una secuencia de codones de ARNm en una secuencia de residuos de
aminoácidos. La información contenida en el ARNm se decodifica a través de la
formación de pares de bases entre secuencias complementarias en los ARN de
transferencia y mensajero. Por lo tanto, al igual que con otros procesos que
implican ácidos nucleicos, la complementariedad entre los pares de bases se
encuentra en el corazón del proceso de traducción. La parte del ARNt que
participa en esta interacción complementaria con el codón del ARNm es un tramo
de tres nucleótidos secuenciales, llamado anticodón, que se encuentra en
el ciclo medio de la molécula de ARNt. Este ciclo está compuesto invariablemente
por siete nucleótidos, los tres medios constituyen el anticodón. El anticodón
se encuentra en un extremo de la molécula de ARNt en forma de L opuesta al
extremo al que está unido el aminoácido.
Es críticamente
importante durante la síntesis de polipéptidos que cada molécula de ARN de
transferencia se una al aminoácido correcto (afín). Los aminoácidos están
unidos covalentemente a los 3 extremos de su (s) ARNt (s) relacionado (s) por
una enzima llamada aminoacil-ARNt sintetasa (aaRS). Aunque hay muchas
excepciones, los organismos típicamente contienen 20 aminoacil-tRNA sintetasas
diferentes, una para cada uno de los 20 aminoácidos incorporados en las
proteínas. Cada una de las sintetasas es capaz de "cargar" todos los
ARNt que son apropiados para ese aminoácido.
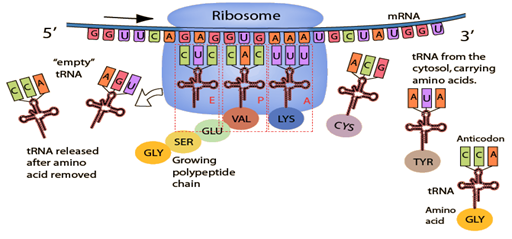
La traducción es el proceso que convierte de ARN mensajero a una proteína. El proceso es mediado por tres tipos de ARN. El ARN mensajero que sirve como mecanismo de transferencia de la información genética, el ARN ribosómicos que reconoce al ARN mensajero y lo encierra en una superestructura llamada ribosoma. Y el a de transferencia, que trasfiere un aminoácido por cada tres bases nitrogenadas. El tipo de aminoácido depende de la secuencia de las tres bases nitrogenadas llamadas codón. Este código ha sido resuelto y por lo general se encuentra expresada didácticamente en la tabla de codones. El ARN de transferencia posee un anticodón que reconoce a su codón, trasfiriendo un aminoácido a la cadena creciente de la proteína. La secuencia sigue creciendo hasta que se llega al codón de parada o hasta que se lee todo el ARN mensajero. Finalmente, la cadena de aminoácidos se enrosca sobre si misma por las interacciones moleculares formando una proteína.
Figura
54. Resumen de la
síntesis de proteínas o traducción genética (YouTube) (YouTube)
Aunque es un muy mal nombre, esta hipótesis ha sido
reforzada por los descubrimientos en biología molecular, desde que fuera
propuesta en los años 70s. En su forma más actual se puede definir cómo: La
información genética fluye desde los ácidos nucleicos hacia las proteínas y
nunca en sentido inverso. Esto implica que no existe un antiribosoma que
pueda transformar la secuencia de aminoácidos en ARN mensajero. Esto explica la
barrera de Weismann sobre la herencia lamarkiana. Si Lamarck hubiera estado en
lo cierto, la célula debería contar con un
A pesar de lo tajante que puede parecer, el dogma central
posee flexibilidades, por ejemplo, no dice nada sobre la direccionalidad del
flujo de información entre los ácidos nucleicos. Aunque originalmente se
consideraba que era direccional (ADN → ARN) la existencia de la enzima ARN
transcriptasa reversa encontrada en retrovirus como el del VIH cambió todo.
Posteriormente los retrotrasposones confirmaron lo que muchos sospechaban ya.
El flujo de información genética entre el ADN y ARN es bidireccional (ADN ↔ ARN)
a tal punto que el genoma humano ha sido esculpido por él. Existen más





No hay comentarios:
Publicar un comentario