(Ciencias de Joseleg) (Biología) (Teoría
de la Biología)
(Genética moderna) (Introducción) (Naturaleza
química del gen) (Conflicto
por la estructura del ADN) (Estructura
del ADN) (La
replicación del ADN) (Síntesis
de proteínas) (Denaturación
y renaturación) (Estructura
del genoma) (Tipos
de mutaciones) (Elementos
móviles del genoma) (Identificación
humana) (Referencias
bibliográficas)
De este
punto en adelante los esquemas para la simbolización del genoma cambian, esto
es debido a la extensión de los genes que, aunque en términos del genoma son
pequeños, en términos absolutos de contar pares de bases pueden ser muy
extensos. Las barras gruesas representan una sección de ADN de interés como un
gen, pero también pueden representar loci
Figura 59. Modelo de barras. Modelo de barras, grueso para lo importante y delgado para lo no
importante de una secuencia de ADN. La secuencia importante o resaltada puede
ser un gen, un fragmento regulador o cualquier otra parte que sea el enfoque de
la investigación.
Debido
a que el ADN es el material genético tendemos a pensar en el como una molécula
estática totalmente conservativa, que o no cambia o lo hace muy poco a través
de la historia evolutiva –eso si es que considera la noción evolutiva en la
biología, pero eso es mas de parte de los no-biólogos. El hecho ineludible es
que el genoma es capaz de realizar cambios muy rápidos, no solo de una generación a la
siguiente, sino al interior de un mismo individuo y más aun de una misma
célula. Estos cambios pueden estar orquestados por mecanismos internos de
origen interno “genéticos” o por la transducción de señales externas
“epigenéticos”. En esta sección veremos como la biología ha adoptado una visión
del mundo como la de Heráclito, todo está en movimiento, en un cambio
constante, la estabilidad –en este caso de las especies y los genomas –es tan
solo una apariencia. Evidentemente esta visión de trasfondo filosófico está
reforzada por pruebas empíricas.
La
primera teoría de la evolución tenía en su punto más débil una explicación
lamarckiana de la genética, y aunque muchos digan que la selección natural es
el motor de la evolución, hay que matizar mucho esa expresión. La selección
natural es un mecanismo ecológico que elimina variantes, no las crea, por lo
que en la base debía existir un mecanismo subyacente, el verdadero motor que
pone en movimiento a la selección natural, aquel motor que arroja las
variaciones que son susceptibles de ser seleccionadas. Años más tarde August
Weismann refutaría la primera teoría de la evolución destruyendo su base
lamarckiana, pero a pesar de que propuso una hipótesis genética de reemplazo
llamada herencia dura, esta no pudo generar una explicación completa del
problema. Posteriormente en 1916 Morgan y colaboradores dan soporte a las
mutaciones como fenómeno que crea nuevos alelos, sin embargo, no sería hasta la
elucidación de la maquinaria molecular y la naturaleza química de los genes que
se pudo tener acceso al conocimiento específico de lo que son exactamente las mutaciones.
En esta serie de artículos veremos la segunda parte de la Teoría Sintética de
la Evolución, aquella que concierne no con la ecología y las poblaciones, sino
aquella que concierne a la genética misma y al verdadero motor de la evolución,
que es la mutación.
Literalmente mutaciones de nucleótido simple son
las más comunes, pero también las menos importantes a la hora de explicar el
origen de los genes, aunque sí que explican su diversificación secundaria. Una mutación de un solo nucleótido es un
cambio en solo un par de bases nitrogenadas sea por inserción, deleción o
cambio. Las inserciones son inocuas a menos que involucren la adición de un
aminoácido crucial para la proteína, pero las deleciones e inserciones pueden
ser particularmente nocivas pues alteran el marco de lectura de una proteína,
lo cual la inhabilita funcionalmente. Los SNP constituyen hasta el 90% de todas
las variaciones genómicas humanas, y aparecen cada 1,300 bases en promedio, a
lo largo del genoma humano. Dos tercios de los SNP corresponden a la
sustitución de una citosina (C) por una timina (T). Estas variaciones en la
secuencia del ADN pueden afectar a la respuesta de los individuos a
enfermedades, bacterias, virus, productos químicos, fármacos, entre muchos
otros, lo cual en últimas conforma mucho del material base con el cual un
individuo se enfrenta a la selección natural. Sin embargo, a pesar de ser tan
comunes, los SNP nos dicen muy poco a cerca de la evolución a largo plazo de
los seres vivos, y en especial en lo que respecta a una pregunta
particularmente difícil si solo se conocen las mutaciones SNP, esa pregunta es
¿Cuál es el origen de los genes?
Figura
60. Las ranas poliploides. El poliploide es más grande, es una tendencia en
las plantas (YouTube), pero que también se observa en las ranas del
género Xenopus spp.
El tipo
de mutación que explica las discrepancias con respecto a una cantidad doble de
cromosomas en especies cercanamente emparentadas se denomina poliploidía. Una
poliploidía se define como un cambio en el material genético en el cual el
genoma completo de la especie es multiplicado por dos, si los parentales poseían
dos cromosomas, los filiales poseerán cuatro cromosomas. La poliploidía emerge
de un error en la meiosis, en la cual los cromosomas no se segregan a los
diferentes gametos, y por lo tanto se producen la mitad de gametos sin
cromosomas y la mitad de gametos con todos los cromosomas, por lo que se
obtiene un gameto diploide. El punto es que se requiere de otro gameto diploide
para que el poliploide se produzca y para esto existen dos opciones. La primera
es que el gameto diploide sea ofrecido por una especie cercanamente emparentada
también por un error en la meiosis. La segunda opción otro individuo de la
misma especie, pero de genero diferente experimente el mismo error y ofrezca el
gameto diploide complementario. Adicionalmente en los animales puede darse una
poliploidía embrionaria en la cual los cromosomas que conforman al cigoto se
duplican, pero no se segregan en células diferentes hasta que una segunda ronda
de duplicación se da, por lo que en el primer clivaje se segregan el doble de
cromosomas de la especie.
El
fenómeno de la poliploidía es particularmente común en las plantas con flor, lo
cual incluye a la mayoría de las especies domesticadas para la producción
agrícola como el trigo, las bananas o el café. Algunas estimaciones afirman que
más del 70% de las especies de plantas con flor son poliploides de otras. La
duplicación de los cromosomas es un evento dramático para un individuo, el cual
puede o no sobrevivir o experimentar cambios drásticos en el modo en que se
relaciona con el ambiente. A simple vista muchos poliploides vegetales son más
grades que sus parientes originales, pero al interior se empiezan a generar
grandes cambios, como la tendencia a la eliminación de los genes redundantes,
mediante el silenciamiento epigénetico o mutacional. El proceso de silenciado
de genes es lento y afecta más a los genes de codifican a las proteínas que los
genes regulatorios. Debido a su lentitud varios estudios han podido reportar la
genética de este proceso en diversos estados de su historia evolutiva. De
cualquier forma, las plantas con flor debido a su naturaleza poliploide tienden
a poseer los porcentajes más altos de ADN codificante en comparación con los
animales. Dado que las plantas con flor son en muchos casos hermafroditas, la
producción de un solo individuo poliploide viable puede conllevar a la
formación de una especie completa, lo cual es un ejemplo de especiación.
Figura
61. Visualizando las duplicaciones. (A) En los
chimpancés, por ejemplo, los
La poliploidía es un caso extreme y dependiendo de la estructura genómica de la especie puede ser o muy común o muy rara. Según los datos recolectados es muy común para las plantas con flor, pero para los demás seres vivos es más bien un proceso extremo y raro. Sin embargo, la poliploidía no es el único mecanismo que le permite a los seres vivos duplicar sus genes, existen formas en que pequeñas fracciones del genoma pueden duplicarse e insertarse de forma tal que adquieren los potenciales que otorgan la compensación de dosis genética. Estudios recienten han demostrado que las duplicaciones parciales de los genes suceden con una frecuencia sorprendentemente alta, lo cual no debería ser sorprendente, ya que la duplicación de los genes es la fuente principal de genes nuevos por medio de la subfuncionalización, la neofuncionalización y la fusión de dominios. Estos procesos son prácticamente uno de los principales motores del proceso evolutivo, ya que sin genes nuevos no habría posibilidad de cambio evolutivo.
Figura
62. Genes y proteínas. Los genes al expresarse forman las proteínas, las
cuales son quienes realizan la mayor parte de las funciones en los seres vivos,
la pérdida de un gen implica la pérdida de una proteína lo cual puede llegar a
ser mortal para el individuo.
De
acuerdo a un estimado reciente, cada gen en el genoma tiene alrededor de un 1%
de probabilidad de ser duplicado cada millón de años, pero si usted posee unos
20 000 genes implica que cada millón de años 200 genes se habrán duplicado. La
duplicación de los genes puede darse por múltiples mecanismos independientes a
la poliploidía entre los que se puede contar los retrotrasposones, la
transferencia viral y la recombinación no equivalente durante la profase I de
la meiosis. Cabe añadir que no todos los genes tienen una misma tasa de
duplicación, algunos tienden a duplicarse más que otros, lo cual implica que
ciertos genes deben presentar una mayor cantidad de familiares en un mismo
genoma.
De
todos los mecanismos se cree que el más común es la recombinación no
equivalente de los cromosomas durante la meiosis, lo cual generaría cromosomas
más largos “con segmentos duplicados o inserción “y más cortos “con segmentos
faltantes o deleción”. La recombinación no equivalente ocurre cuando los
cromosomas que se encuentran en profase 1 no se alinean perfectamente. Los individuos
que reciben los cromosomas con deleciones pueden sufrir patologías graves o
incluso morir, pero en el caso de la inserción la compensación de dosis puede
permitir al cromosoma nuevo no afectar mortalmente a su portador.
Otra consecuencia de esto, es que cuando el cromosoma alargado se recombine en la siguiente generación, los genes duplicados se intercambiarán en una cantidad diferente, o lo que es lo mismo, se genera una diversidad en los genes en la región afectada por la inserción, lo que conlleva a la generación de un nido de genes duplicados. Esta variación en el número de copias de un gen al interior de un nido “cluster” ha sido reportada para genes como el de la amilasa en el ser humano, en el cual se ha reportado variación entre 4 y 10 copias. Lo anterior implica que los cromosomas homólogos no necesariamente portan exactamente la misma información, tan solo requieren una integridad mínima para reconocerse mutuamente durante la meiosis, pero existe cierta flexibilidad en sus tamaños y contenidos. A esta tolerancia al cambio en sistemas complejos e integrados se la denomina resilencia.
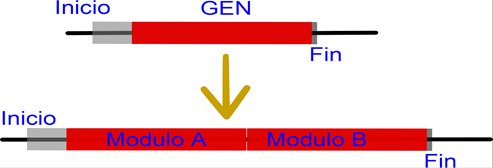
Figura
63. Los genes son multi-modulares. Los genes pueden ser expresados en términos de
módulos, sean estos repetitivos o no. Existe otro tipo de modularidad que se
llama estructura cuaternaria, en el cual los módulos se encuentran separados
como si fueran loci independientes.
Un
pequeño porcentaje los genes que se encuentran en proceso de perderse en el
caos del genoma no codificante adquieren mutaciones que otorgan ventajas a sus
portadores mediante la adquisición de nuevas funciones. Cuando un gen lleva a
cabo el proceso de neofuncionalización y subfuncioanlización apoyado por la
compensación de dosis de una copia de respaldo se la denomina isoforma. Para
ilustrar estos procesos analizaremos la familia de genes de las globinas,
aunque ya lo hemos hecho con la cascada de coagulación
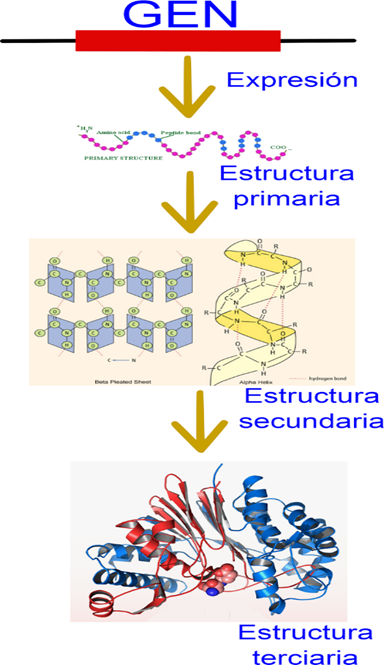
Aunque
por lo general se nos dice que las proteínas tienen cuatro niveles de
complejidad:
·
Secuencia
lineal o estructura primaria, esta se genera cuando el ribosoma la fábrica a
partir de los mensajeros de ARN.
·
La
estructura secundaria es una conformación que adquiere la secuencia de
aminoácidos cuando las interacciones moleculares empiezan a afectarlas
generando hélices o láminas.
· La estructura terciara es una proteína funcional compuesta de hélices y láminas.
Figura
64. Defuncionalización deletérea. La defuncionalización es un proceso común, cuando
un gen vital pierde su función, el indivioduo muere en el embrión o se genera
una enfermedad en cualquier momento de la vida, disminuyendo la aptitud
darwiniana del individuo.
Sin
embargo, está visión es engañosa, entre la estructura secundaria y la terciaria
podemos ubicar otro nivel de complejidad que podemos llamar dominios modulares. Una proteína funcional puede estar compuesta
por varios dominios funcionales, los cuales le permiten operar en un
determinado contexto.
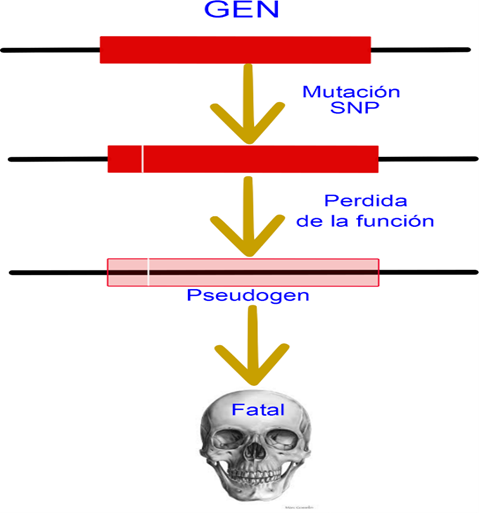
La
defuncionalización hace referencia a la pérdida total del gen, ya sea porque la
proteína generada es no funcional –lo cual genera fenotipos recesivos. También puede darse el caso de que el
mensajero de ARN al haber mutado no pueda ser leído por el ribosoma, o incluso
que las enzimas que leen al ADN no puedan generar el mensajero de ARN, a esto
se lo denomina cambio en el marco de lectura –efectos conocidos de mutaciones
estilo SNP.
A parte de esto existen cambios epigenéticos que impiden que un gen duplicado se exprese, por ejemplo, las histonas que son proteínas estructurales del genoma pueden enroscarse en torno a las secciones duplicadas impidiendo su lectura por las enzimas especializadas. En otros casos, el gen mismo puede metilarse, lo cual impide que las enzimas puedan leerlo.
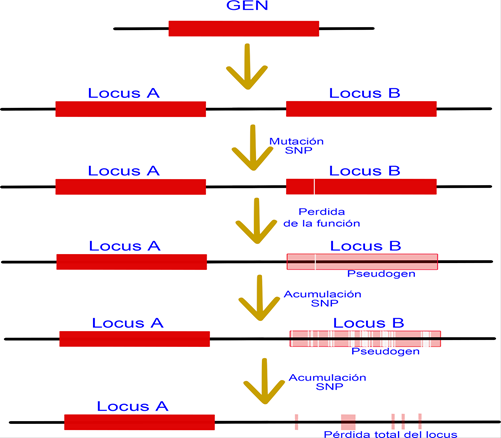
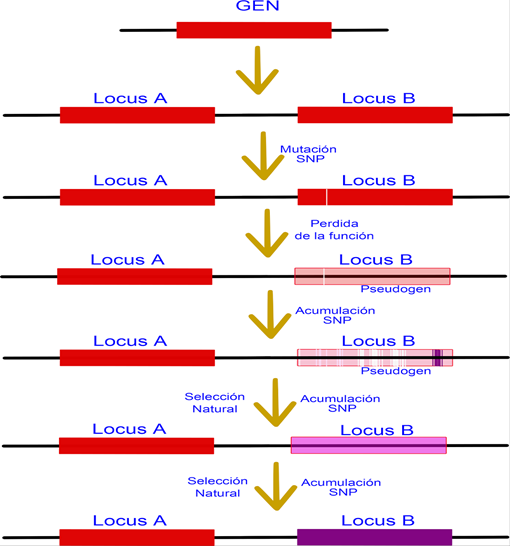
Figura 65. Defuncionalización no deletérea. Este es el destino más común para los genes duplicados, mientras que un locus es activo y evita que el individuo muera, con las generaciones el otro locus acumula mutaciones hasta perderse en el caos del ADN no codificante. Aun así, con las adecuadas técnicas de secuenciación es posible encontrar los rastros de estos genes perdidos o pseudogenes.
Figura
66. Subfuncionalización. La subfuincionalización, permite especializar los
loci, lo cual puede ser conveniente a los seres vivos cuando se exponen a
nuevos retos ambientales.
Si
dicho gen se perdiera por mutaciones del tipo SNP o por epi-alteraciones como
el cambio de las histonas o la metilación del ADN entonces el organismo podría
verse en graves riesgos de morir por defectos energéticos. Un gen duplicado
permite que aun cuando uno de los nuevos
La
pérdida de la función de una proteína no necesariamente afecta a todos sus
dominios funcionales, como mencionamos anteriormente, si al interior de un
mismo gen se encuentran regiones funcionales, la pérdida de una de sus
funciones puede conllevar a que dicho gen evolucione para realizar
específicamente la función o funciones que le han quedado. Otro tipo de subfuncionalización puede
abarcar la reducción de sustratos sobre los cuales actúa una proteína. En
cualquier caso, todos estos fenómenos se basan en mutaciones de tipo SNP o en
mutaciones en bucle o tándem.
Figura
67. Fusión de módulos genéticos. Una vez que los dos módulos quedan conectados en
un solo producto génico, uno de ellos puede acumular SNP sin riesgo de
selección negativa para su portador. Con el tiempo uno de los módulos puede
evolucionar a la perdida de la función, con lo que se convertiste en un
elemento estructural de la proteína o a una nueva función, generando una
proteína con múltiples dominios funcionales.
Bueno
¿y cómo aparece una proteína multimodular en primera instancia?, bueno esto no
ocurre en las mutaciones de genoma completo, pero lo explicaremos aquí de todas
formas. Por lo general los genes están flanqueados por secuencias regulatorias
y un iniciador de la secuencia, así como un finalizador, el cual le dice a las
enzimas especializadas –inicie a leer aquí y termine de leer aquí.
Si una
mutación corte de forma tal que el segmento duplicado queda en tándem con
respecto al viejo borrando la sección de terminación, la enzima leerá las dos
secciones generando un único mensajero. Este mensajero único al ser leído por
el ribosoma creará una proteína el doble de grande con dos dominios exactamente
iguales. Lo cual permitirá que uno de ellos pueda evolucionar a funciones
nuevas mientras que el viejo sigue su función normal.
Un gen
que está bajo compensación de dosis está libre de selección natural, lo cual le
permite acumular rápidamente mutaciones por deriva genética. En muchos casos
puede llevar a la perdida de la función y convertirse en un pseudogen, pero en
otras puede ocurrir algo diferente.
La generación de funciones nuevas no necesita realmente grandes cambios, tal vez solo la alteración de una reacción química a otra muy relacionada altere la acumulación de un compuesto flexible por uno duro, en tal caso el organismo pasa de ser flexible y vulnerable, a inflexible pero muy duro. En cualquier caso, la nefuncionalización hace referencia a la adquisición de funciones aparentemente nuevas de un gen duplicado.
Figura
68. Neofuncionalización. Los pseudogenes al estar libres de la selección
negativa pueden en ocasiones adquirir funciones nuevas que otorgan a sus
portadores leves ventajas con respecto a sus competidores, lo cual los
selecciona positivamente, permitiendo que acumulen mutaciones que filtradas por
la selección conllevan a la formación de nuevas funciones y al nacimiento de
nuevos genes.
Evidentemente
en el caso del cambio genómico una proteína puede tener simultáneamente en su
historia duplicaciones, deleciones, subfuncionalizaciones solo para volver a
duplicarse y quedar encadenada de nuevo formando nuevamente una proteína con
más módulos.
Es por
esta razón que el análisis evolutivo de las proteínas es tan complejo, pues
pueden encontrarse rastros de módulos funcionales en proteínas que no se
esperaría. Sin embargo, estos rastros conforman la base de una rica historia
evolutiva que puede ser interpretada y que sirven como base para la generación
de modelos evolutivos para sistemas que son muy complejos.
A pesar
de lo complejo de este relato la propia secuenciación del ADN la ha
corroborado, por todo el genoma se encuentran los restos de genes relacionados
con otros funcionales, en el interior de genes funcionales se pueden encontrar
restos de módulos no funcionales que sí lo son en otras proteínas.






No hay comentarios:
Publicar un comentario