(Ciencias de Joseleg) (Biología) (Teoría de la Biología) (Genética moderna) (Introducción) (Naturaleza química del gen) (Conflicto por la estructura del ADN) (Estructura del ADN) (La replicación del ADN) (Síntesis de proteínas) (Denaturación y renaturación) (Estructura del genoma) (Tipos de mutaciones) (Elementos móviles del genoma) (Identificación humana) (Referencias bibliográficas)
La reproducción es una de las propiedades más importantes de los seres vivos, a tal punto que no existe definición de ser vivo que no la toma en consideración como una de las propiedades sine qua non que debe poseer cualquier sistema para ser designado como vivo. Inicialmente el proceso de reproducción es estudiado a nivel estructural y biológico, sin embargo, durante la etapa de síntesis en el ciclo celular se da otra historia, una historia de índole bioquímico. La célula duplica la concentración de ADN en el núcleo empleando una serie de proteínas especializadas, las cuales también están involucradas en la reparación del genoma, lo cual reduce al máximo la cantidad de mutaciones posibles, pero no las elimina por completo.
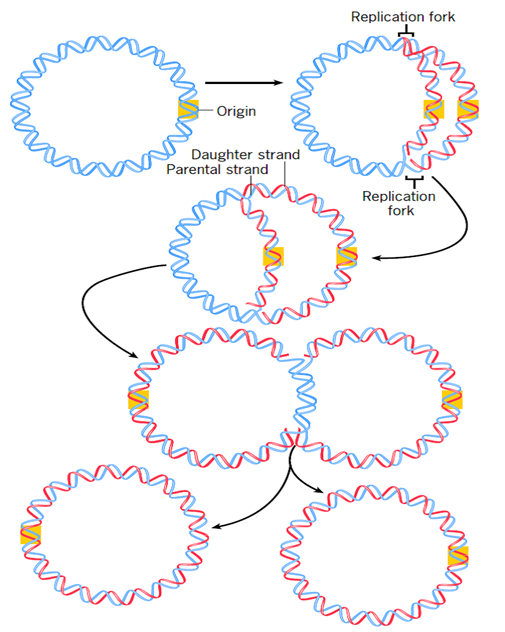
Figura
25. Replicación del ADN. Acercamiento a la propuesta original para la
replicación del ADN, cada una de las hebras antiguas "azul" sirven
como templado para la creación de la nueva "anaranjado", haciendo del
proceso algo semiconservativo.
El modelo propuesto por Watson y Crick basado en
las difracciones de rayos X realizadas por Rosalind Franklin en 1953 fue
acompañada por una sugerencia para un mecanismo de replicación autodirigido.
Las dos hebras de la molécula se mantienen juntas mediante puentes de hidrógeno
generados por las bases nitrogenadas, dos para la pareja adenina-timina y tres
para la pareja citosina-guanina. El rompimiento de estos puentes de hidrógeno
podía realizarse por temperatura in vitro, pero in vivo debía involucrar
una batería de enzimas especializadas. Una vez que las dos hebras estuvieran
separadas, cada una serviría como patrón o templado para la formación de una
hebra nueva.
El modelo de Watson y Crick realiza ciertas predicciones muy específicas con respecto al comportamiento del ADN durante su replicación en la fase de síntesis del ciclo celular. De acuerdo con su propuesta, cada una de las moléculas hijas posee una hebra madre original heredada y una hebra hija formada a partir de la hebra madre que sirve como templado. A este tipo de replicación se la denominaría semiconservativa. Evidentemente esta no era la única hipótesis en su tiempo a cerca de la replicación del ADN.
Figura
26. Hipótesis para la replicación del ADN. "Izquierda" modelo de replicación
semiconservativa durante dos generaciones. "Medio" modelo de
replicación conservativa durante dos generaciones. "Derecha" modelo
de replicación disruptiva durante dos generaciones.
En la replicación conservativa, las dos hebras originales se volverían a juntar, de modo tal que crearía dos hebras hijas que a su vez se juntaría, de este modo se crearía una molécula de doble hélice hija y una molécula de doble hélice madre. En la hipótesis de la replicación dispersa, las hebras de la molécula madre se romperían en fragmentos, y la nueva molécula se sintetizaría a partir de estos fragmentos cortos. Los fragmentos viejos y los nuevos serían entonces reensamblados al azar, generando así una mezcla de ADN parental y ADN recientemente sintetizado.
Figura
27. Marca radioactiva/masa de nitrógeno. Los símbolos y colores arbitrarios para la cadena
pesada "izquierda" y ligera "derecha" asociadas al
nitrógeno 15 y nitrógeno 14 respectivamente. Dado que el marcador es el
nitrógeno, la diferencia entre las dos hebras de ADN se ubica en las bases
nitrogenadas
Aunque la replicación disruptiva se veía como poco probable, la única forma de refutar los modelos era poder distinguir entre las secuencias madre y loas secuencias hijas en una replicación controlada. El método para lograr esto fue desarrollado en 1957 por Matthew Meselson "1930-vivo" y Franklin Stahl "1929-vivo" del Caltec empleando isótopos pesados de nitrógeno 15 e isótopos ligeros de nitrógeno 14. Los diferentes isótopos ayudarían a distinguir entre las hebras madre e hija. El principio es la densidad, una molécula que contiene únicamente nitrógeno 15 es más densa, mientras que otra que contiene solo nitrógeno 14 será menos densa. La relación de densidad intermedia será de números fijos en el caso de una replicación semiconservativa, o de una variación continua entre los dos extremos en el caso de una replicación disruptiva al azar.
Figura
28. Efecto macroscópico en centrífuga. Al centrifugar los blancos se obtienen dos fases
"invisibles", una para las moléculas densas y otra para las moléculas
ligeras. La centrifugación es un proceso
que sirve para separar sustancias por su densidad, en el ejemplo de la sangre,
los glóbulos rojos tienden al fondo por su alto contenido de hierro, en medio
quedan los glóbulos blancos, y en la parte superior queda la solución líquida
llamada plasma. El proceso funciona también para el ADN (YouTube).
Este
experimento será uno de los experimentos cruciales en la historia de la
biología molecular. Sus resultados permitirían corroborar que la propuesta de
replicación generada por Watson y Crick junto con su modelo de la doble hélice
era correcta. Hay que rescatar que los biólogos moleculares no poseen
microcamaritas para ver el genoma, por lo que en su contexto cualquier
hipótesis sobre la replicación del ADN podría ser correcta. Las hipótesis a
manejar eran tres: replicación semiconservativa, replicación conservativa y
replicación disruptiva. La metodología empleada fue mediante isótopos marcados,
la medición se realizaría por la densidad de la molécula resultante.
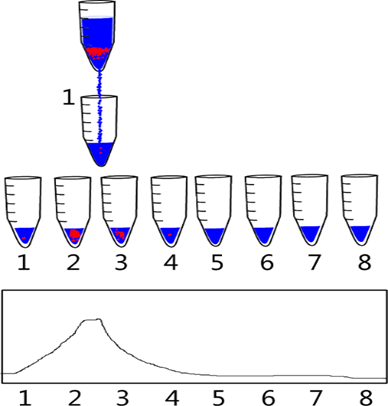
Primero
que todo hay que crear el patrón para la molécula de ADN de alta densidad, para
esto simplemente se hace crecer una colonia de bacterias en un medio que solo
contiene nitrógeno 15. Con el tiempo, la colonia solo puede integrar nitrógeno
15 en su nuevo ADN. De esta muestra se toma una parte para la determinación de
la densidad y otra para realizar la fase experimental. Por otra parte, se
tendría un cultivo de bacterias que crecen en un medio que contiene solo
nitrógeno 14, este cultivo proporcionará la muestra para determinar la densidad
de una molécula de ADN que contiene solo nitrógeno 14. Con los valores de los
extremos, muy denso y poco denso puede crearse una escala con las muestras
experimentales en los siguientes pasos.
A
B
C
Figura 29. Experimento de Meselson-Stahl. (A) Cada
uno de los blancos arrojan picos únicos en los extremos de la tabla. (B) La espectrofotometría arroja valores con los
cuales se puede construir una gráfica que indica la cantidad de sustancia en
una solución. Al decantar el tubo centrifugado en varios tubos, estos generan
un gradiente de concentraciones. A estos tubos se les extraen muestras para el
espectrofotómetro y así obtener las concentraciones. (C) Este es el resultado más cercano, dos picos
uno para las moléculas conservadas y otro para las nuevas, sin intermedios de
ninguna clase.
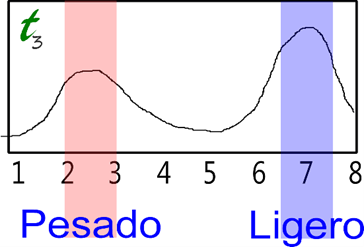
La determinación de la densidad fue realizada
mediante la centrifugación de equilibrio o centrifugación isopicnica. En este
procedimiento la muestra de ADN sometida a la centrifugación se mueve de forma
diferencial en el tubo. La más densa termina en el fondo y la más ligera
termina cerca de la superficie. En
el experimento Meselson-Stahl, el blanco de ADN con nitrógeno pesado termino
más al fondo, mientras que la muestra que había sido incubada con medio de
nitrógeno ligero terminó más cerca de la superficie. En medio de ambas se pudo
crear una escala, entre más cercana fuera la molécula a la forma perfectamente
pesada, esta migraría al fondo, y entre más cercana fuera a la forma
perfectamente ligera esta quedaría cerca de la superficie.
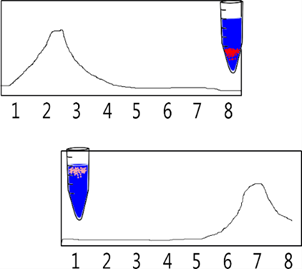
Bueno,
usted centrifuga, pero lo que sale de la centrífuga es un fluido transparente,
el problema es ver lo que es invisible al ojo humano. Para esto, se pasa el
fluido del tubo de centrifuga a otros tubos en orden como se muestra en Figura 29-A. Para observar lo que no
puede ser visible se emplea el método de espectrofotometría, la cual arroja
picos dependiendo del tubo de muestra que has obtenido al decantar el tubo de
centrifugación. De esta forma cada una de las muestras puras arrojará picos
semejantes a estos Figura 29-B. En el modelo conservativo se
esperaría la formación de dos bandas al final del procedimiento, una para las
hebras pesadas y otra para las hebras ligeras. Cada pico corresponde a las
hebras originales con las que inició el cultivo y a las hebras hijas que son
completamente nuevas y ligeras Figura 29-C.
El
modelo disruptivo propone la destrucción de las cadenas de la primera
generación y la conformación de moléculas hibridas en fragmentos, con el paso
de las generaciones las moléculas integraran más y más del nitrógeno ligero. En
el modelo semiconservativo durante la primera generación, todas las moléculas
pesadas son convertidas a moléculas híbridas, y en las siguientes generaciones,
aunque se producen más moléculas ligeras, siempre se conserva una cantidad de
moléculas hibridas. Los resultados del experimento arrojaron la formación del
pico esperado para la molécula híbrida, es decir, corroboraron los resultados
esperables si la molécula realizara la replicación de forma semiconservativa.
Para 1960 pudo demostrarse lo mismo en el modelo biológico de los eucariotas,
por lo que, en el ser humano, la molécula de ADN también se realiza la
replicación de ADN de forma semiconservativa. El procedimiento experimental
involucraría el uso de un marcador medible mediante microfotografía de los
cromosomas.
Aunque suene un poco antropocéntrico, en términos cualitativos y cuantitativos, el ADN de las bacterias se encuentra organizado de un modo más sencillo que en los eucariotas, por esta razón fue primero en las bacterias donde se pudo estudiar el modo en que el ADN se replica. Específicamente, el modelo biológico fue Escherichia coli, la bacteria más conocida por la ciencia y de quien es posible que se tenga una leyenda negra. Igual que sucede con la mayoría de las bacterias, E. coli posee una enorme diversidad de variantes a las cuales llamamos cepas, algunas cepas son muy peligrosas e incluso hasta fatales como E. coli O157H7, pero las cepas que se emplean en el laboratorio de biología molecular son inocuas. Y no es que una bacteria inocua genere por arte de magia todos los mecanismos de patogenicidad necesarios para hacerse infecciosa en unas cuantas generaciones, el empleo de E. coli es completamente normal y seguro en laboratorios de bajos niveles de bioseguridad.
Figura
30. Cultivo bacteriano. A una temperatura restrictiva un mutante
termosensible no podría generar colonias como las que se ven en la imagen,
aunque la bacteria esté allí no puede desarrollarse.
Figura
31. Cultivando bacterias. En este contexto, in vitro se refiere a que las
proteínas y los procesos se llevan a cabo fuera de las células, mientras que in vitro se refiere al proceso
que se lleva a cabo al interior de la célula.
Dos aproximaciones experimentales serían cruciales para el entendimiento de la replicación del ADN en las bacterias, la primera es el empleo de mutantes sensibles y la otra es el de la replicación in vitro. La existencia de cepas de laboratorio con mutaciones que impiden la síntesis de una o varias de las enzimas necesarias para la replicación. Aunque esto pueda parecer extraño, el aislamiento de mutantes su cromosoma solo es posible si la mutación es sensible a la temperatura, lo cual permite el majeo de ambos fenotipos. Cuando necesitas hacer crecer el cultivo lo haces a una temperatura permisiva, y cuando requieres ver el efecto de la mutación, haces crecer el cultivo a una temperatura restrictiva. Los mutantes sensibles a la temperatura han sido aislados para virtualmente cualquier actividad fisiológica, y han sido particularmente importantes para el estudio de la síntesis de ADN durante la replicación, la reparación y la recombinación genética.
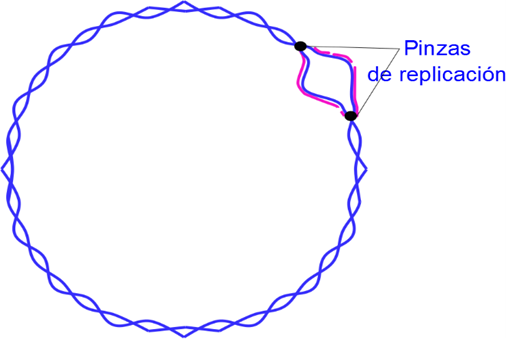
Figura
32. Pinzas de replicación. Las pinzas de replicación en un cromosoma
bacteriano, abren el ADN para que las proteínas asociadas inicien la copia y
síntesis de nuevo ADN.
El desarrollo de sistemas in vitro en
el que la replicación puede ser estudiada empleando componentes extraídos y
purificados de la célula. En algunos estudios la molécula de ADN es incubada
con extractos celulares en los que las proteínas sospechosas de tener algo que
ver con la replicación son empleadas o removidas, confirmando resultados con
los mutantes termosensibles. Tomados en conjunto, estas aproximaciones
experimentales han revelado la actividad de más de 30 proteínas diferentes
necesarias para la replicación del ADN en Escherichia coli. En los siguientes artículos discutiremos estos
procesos en la medida en que han sido aclarados algunos, pero otros aún siguen
en investigación.
Aunque es totalmente merecido que se designe a la
molécula del ADN como la molécula de la vida, si es necesario recalcar que, por
sí mismo el ADN está tan vivo como cualquier polímero plástico sintético que se
convierte en fibras transparentes en un vaso de precipitado. El ADN por sí
solo, desnudo no está vivo, no puede realizar ninguna de las funciones que
asociamos con la vida, es necesaria la presencia de una enorme batería de
proteínas, es decir, de la célula, para que este pueda replicar, producir y
mutar. Esta aparente dicotomía entre la función del ADN y la función de las
proteínas conllevaría a la definición del problema del huevo y la gallina en el
origen de la vida. Sin embargo, para nuestro contexto, no discutiremos aun este
problema.
El cromosoma de las bacterias es circular, esto condiciona mucho de la maquinaria molecular empleada para replicarlo. La replicación inicia en un sitio específico del cromosoma bacteriano, este lugar se denomina origen de replicación. El origen de replicación en Escherichia coli es una secuencia específica llamada oriC donde varias proteínas se ensamblan para iniciar el proceso de replicación. Una vez iniciado, el proceso se da en ambas direcciones opuestas, lo cual se conoce como bidirecionalidad. Los sitios donde los segmentos replicados se unen con el ADN no replicado se denominan pinzas de replicación. Cada pinza de replicación corresponde al sitio donde la doble hélice parental está siendo separada y donde los nucleótidos están siendo incorporados.
Figura
33. Replicación de un cromosoma bacteriano. Cada una de las pinzas de replicación sintetiza
ADN, para que el cromosoma se duplique, el proceso es tan rápido que no se ha
terminado de completar el cromosoma nuevo cuando ya inicia su propia
replicación.
Las dos pinzas de replicación se mueven en
direcciones opuestas hasta que se encuentran en el punto opuesto del origen de
replicación. En este punto, los dos cromosomas se separan y migran a los polos
opuestos de las células. Es importante resaltar que debido a la peculiaridad
del modo de vida de Escherichia coli,
la replicación del cromosoma dará inicio aun antes de que las dos células hijas
se hallan separado. Esto hace de la replicación bacteriana algo muy rápido, ya
que la fase de síntesis y de crecimiento se da de forma simultánea.
Para que el ADN pueda ser replicado o leído, las
hebras de ADN deben estar relajadas y liberadas en una configuración simple y
lineal. Cuando las dos hebras de la doble hélice de un cromosoma circular
empiezan a separarse ocurre algo que es muy común para cualquiera que trata de
lidiar con cuerdas o fibras y es el superenrollamiento. En resumen, la
separación de las dos hebras empieza a generar una tensión de torsión en las
zonas del cromosoma que aún no han sido separado generando un
superenrollamiento positivo. A esto se lo denomina relajación “unwinding” de la
doble hélice.
A diferencia de una cuerda que puede superenrollarse, el ruperenrollamiento del ADN provocaría la obstrucción de la replicación a medida que las pinzas de replicación se aproximan a los nudos de mayor tensión. Considerando que un cromosoma bacteriano de Escherichia coli puede generar en potencia 400.000 nudos, y que estos deben ser resueltos en menos de 40 minutos, la magnitud de este problema hace ver al nudo gordiano como un juego de niños. ¿Cómo resolver este problema topológico en un marco de tiempo entre minutos y segundos? La respuesta es el empleo de las topoisomerasas de tipo II.
Figura
34. Dirección de replicación. Originalmente se proponía que ambas hebras
crecían de forma continua en dirección a la pinza de replicación.
Las enzimas especializadas en aliviar las tenciones
de torsión en el superenrollamiento del ADN son las tipoisomerasas de tipo II,
específicamente para la replicación, la enzima es denominada ADN girasa, la
cual como cualquier topoisomerasa de tipo II corta el ADN permitiendo a la
hebra que genera la tención girar, luego esta misma enzima repara la hebra
cortada. La ADN girasa viaja a lo largo de la cabeza de la pinza de
replicación, removiendo los nudos “superenrollamientos positivos”, el proceso
requiere sacrificar energía en forma de ATP. En los eucariotas el mecanismo es
similar.
Figura
35. Arthur Kornberg. (Brooklyn, Nueva York; 3 de marzo de
1918-Stanford, California; 26 de octubre de 2007) fue un bioquímico
estadounidense. Estudió medicina en la Universidad de Rochester, donde se doctoró
en 1941. Permaneció trabajando en el Servicio de Salud Pública de Estados
Unidos durante diez años. En 1952 fue nombrado jefe del Departamento de
Microbiología de la Universidad Washington en San Luis, posteriormente aceptó
la plaza de jefe del Departamento de Bioquímica de la Universidad Stanford, de
California.
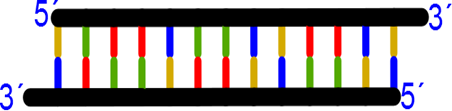
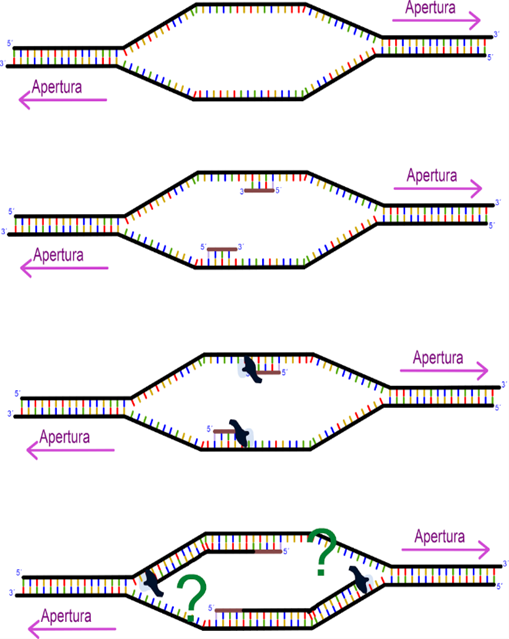
La molécula de ADN en el modelo aceptado posee dos hebras que son antiparalelas y se designan en base al carbono que ceda libre en el azúcar en cada punta. Si una de las hebras inicia en el extremo 3´ entonces terminará en el extremo 5´. De forma antiparalela la hebra complementaria debe comenzar en el extremo 5´y terminar en el 3´- Los sistemas biológicos tienen una serie de complicaciones extrañas, por ejemplo, uno esperaría que la enzima que duplica al ADN posea dos versiones también antiparalelas, mientras que una cataliza la copia desde 3´→ 5´, la otra copiaría la versión antiparalela desde 5´→3´. Sin embargo, en la realidad esto no es tan fácil.
Figura
36. Modelo de peine. Cuando dos peines se reconocen forman la escalera,
que es una analogía más común para la estructura del ADN
El estudio de las enzimas que se encargan de copiar al ADN comenzó en la década de los 50s, básicamente cuando la comunidad científica acepto que (1) la molécula de ADN era la naturaleza química del gen y que (2) existiera un modelo para la estructura química de dicha molécula, el problema a resolver fue como es que esta molécula logra copiarse a sí misma. En la misma década en que estos dos conceptos se aceptaron “1950-1960”, Arthur Kornberg “1918-2007” de la Universidad de Washington dio inició al estudio de las enzimas replicativas. En sus estudios iniciales, Kornberg y colaboradores purificaron una enzima de extractos bacterianos, esta enzima era capaz de incorporar nutrientes con elementos marcados radioactivamente, los cuales eran empleados para construir nuevo ADN. Esta enzima fue denominada polimerasa de ADN, aunque años después será renombrada como la ADN polimerasa I.
Figura
37. Separación del peine. Cuando cada hebra está separada, cada una forma un
peine.
Para que la síntesis de ADN pudiera ser llevada a
cabo por la ADN polimerasa I, la enzima requiere de todas las cuatro bases
nitrogenadas, de lo contrario la copia se detiene. Esto implica que la enzima
agrega de una en una cada base y cuando faltan se detiene en el ensamblado. Si
todo sale bien, las hebras marcadas radioactivamente poseían la misma
composición en términos de porcentajes de bases nitrogenadas que las secuencias
parentales, lo cual sugería que las secuencias madre servían como templado de
las secuencias hijas.
Si bien admiramos el modelo de Watson y Crick para
el ADN, a la hora de visualizar la función de los genes o como en este caso la
replicación, dicho modelo se hace muy complejo. Es por esto que es bastante
común en los libros de biología molecular expresar al ADN mediante modelos
simplificados. El modelo empleado para representar la molécula de ADN en la
replicación y la transcripción es el modelo de peine. Se lo llama así porque el
esqueleto de la molécula es representado como una cinta, mientras que las bases
nitrogenadas se representan como las cerdas del peine.
Analicemos un poco más sobre esto, dado que la secuencia hija es generada a través del copiado de la secuencia madre, la similitud entre la secuencia de dos moléculas de ADN establece una relación directa de parentesco. Las enzimas no son perfectas y como veremos posteriormente poseen un ritmo de errores, lo cual conlleva a que la molécula que se copia será semejante, pero no exactamente igual a su molécula madre. De lo anterior se concluye que entre menos parentesco tienen dos moléculas de ADN el nivel de similitud entre sus secuencias disminuye cuantitativamente. Este razonamiento será de vital importancia para la genética evolutiva, pues si las especies están relacionadas de forma filial ancestro-descendiente, sus secuencias de ADN deben poseer grandes niveles de similitud entre sí. Esta es una predicción sobre la que volveremos en artículos futuros.
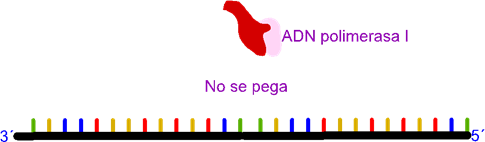
Figura
38. Importancia del cebador. Sin cebador la polimerasa de ADN no se acopla al
templado para iniciar el copiado.
Como vimos anteriormente, el cromosoma bacteriano
se relaja cuando inicia la replicación, esto se debe a que si la ADN polimerasa
interactúa con una molécula de doble hebra, no ocurre nada. Esto fue un
resultado inesperado para los primeros biólogos moleculares. En conclusión,
para que la ADN polimerasa pueda iniciar la replicación es absolutamente
necesario que la molécula esté relajada, es decir, que las dos hebras del ADN
se encuentren separadas y en una disposición lineal.
Lo que fue verdaderamente extraño es que la ADN
polimerasa junto con una molécula de una sola hebra de ADN tampoco hace nada
(…), pero cuando se adicionaban fragmentos de ADN muy cortos “unos 20
nucleótidos” a la mezcla, inmediatamente comenzaba el proceso de replicación.
Ni una molécula de doble hebra, ni otra molécula de hebra simple son capaces de iniciar la replicación. Se requiere de una hebra relajada sobre la cual se posiciona un segmento de ADN muy corta, sobre esta punta es que la polimerasa de ADN se posiciona, en el extremo 3´ y comienza a adicionar nucleótidos, alargando la molécula siempre por la punta 3´. Dado que el segmento corto de ADN es el que da el punto de inicio para la copia se lo ha denominado cebador, secuencia iniciadora o primer para la copia del ADN.
Figura
39. Copiado dirección 5→3. Acoplado el cebador, la ADN polimerasa inicia la
síntesis de la cadena complementaria nueva.
Todas las polimerasas de ADN sean procariotas o
eucariotas tienen los mismos requisitos básicos: (1) Una molécula de ADN
relajada que exponga cada una de las hebras de forma lineal, esta servirá como
el original o templado a partir del cual se generará la copia; (2) Un fragmento cebador o primer de ADN que permite
a la polimerasa adicionarse en el extremo 3´ e iniciar la adición de
nucleótidos. Estos requisitos explican porque ciertas estructuras de ADN fallan
en promover la síntesis del ADN. Una molécula de doble hebra no hace nada
porque no expone el templado, una molécula de hebra simple perfecta tampoco
hace nada porque carece del primer. Una molécula de hebra simple con pequeños
segmentos dobles permite que las polimerasas se peguen en el extremo 3´e
inicien el copiado. Sin embargo, el descubrimiento de los cebadores in vitro
levantó la pregunta ¿Cómo ocurre el proceso in vivo?
La ADN polimerasa purificada por el grupo de investigación de Kornberg identificó una propiedad limitante en la polimerasa de ADN, la cual sería luego encontrada en las demás. Las polimerasas de ADN solo pueden pegarse en la punta 3´ del primer, y toma a los nucleótidos pegando el extremo 5´, y al avanzar sobre el nucleótido nuevo vuelve a posicionarse en el extremo 3´. Lo anterior implica que las moléculas de ADN se encuentran limitadas a sintetizar solo en la dirección 5´→ 3´.
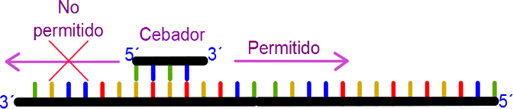
Figura
40. Dirección no permitida. Las polimerasas de ADN son unidireccionales,
pueden copiar de 5→3, pero no de 3→5.
En un claro contraste a lo que propusieron
originalmente Watson y Crick, no existe versión de la ADN polimerasa que
catalice la síntesis de ADN en la dirección 3´→ 5´ lo cual complica el
mecanismo de replicación de la hebra de ADN de la misma dirección. En el
diagrama de replicación del cromosoma circular se esperaría que ambas hebras se
sinteticen de forma continua, esto es fácil para la cadena 5 ´→ 3´, pero la
otra hebra presentará dificultades. Por el momento llamaremos a la cadena de
fácil replicación como hebra líder o cadena líder, mientras que la cadena de
difícil replicación será denominada hebra rezagada o cadena rezagada. Sin
embargo, los resultados de Kornberg aún no refutaban la existencia de la
hipotética enzima que cataliza en la hebra rezagada de forma antiparalela, en
su contexto los biólogos moleculares aún se preguntaban por la existencia de
otras polimerasas de ADN, y por la posibilidad de que, en el contexto celular,
el proceso ocurriera de una forma diferente.
Durante la década de 1960-1970 se dieron pistas de
que la polimerasa de Kronberg no era ni la púnica ni la más importante
polimerasa de ADN. En 1969 la pista fundamental para dicha aseveración fue el aislamiento
de un mutante de Escherichia coli que
tenía una mutación que inhabilitaba la función de la polimerasa de Kronberg en
casi un 99% y aun así, la bacteria era capaz de replicarse a una velocidad
normal. Estudios posteriores revelaron la existencia de otras poliomerasas de
ADN, por lo que la polimerasa de Kronberg fue denominada ADN polimerasa I.
Tiempo después se encontró que la polimerasa que
contribuía principalmente a la replicación del ADN fue la tercera en ser
descubierta, es decir, la ADN polimerasa III. Una bacteria tipia posee entre
300 y 400 moléculas de ADN polimerasa I pero solo 10 copias de la ADN
polimerasa III. En otras palabras, aun cuando su efecto es más potente, pasaba
de ser percibida por la ADN polimerasa I. Sin embargo, ninguna polimerasa que
se iba descubriendo fue capaz de resolver el enigma, todas se pegaban
exclusivamente al extreme 3´de cebador.
El problema con el que se encontraron los biólogos
moleculares a la hora de determinar la función de las polimerasas de ADN es que
todas sintetizan al pegarse en el extremo 3´de un cebador o primer de ADN. Esto
traía consecuencias sobre el modo de replicación propuesto por Watson y Crick
que presuponía la existencia de una ADN polimerasa que podía pegarse al extremo
5´y copiar.
los cebadores son secuencias de ADN, pero la
pregunta surgía, ¿Cómo podía crearse el cebador si ninguna polimerasa de ADN
podía crearlo en primera instancia? Los cebadores al ser ADN requerirían una
polimerasa de ADN, pero las polimerasas de ADN solo actúan cuando hay un
cebador presente, lo cual nos trae a un dilema de tipo huevo gallina. En la
siguiente serie de artículos veremos cómo es que estos dilemas fueron resueltos
experimentalmente.
La falta de polimerización de las polimerasas de ADN en la dirección 3´→ 5´posee una explicación bastante simple, es imposible químicamente, eso es todo. Los procesos biológicos son en últimas al nivel moléculas, procesos que dependen de las leyes de la química. Todo está en la química del carbono 5 y el carbono 3 en la desoxirribosa. Ambos carbonos portan el grupo fosfato, pero a diferencia del carbono 5´, el 3´no puede girar, y esta posición facilita un ataque nucleofílico por parte del nucleótido que ingresa a la cadena. Esta estabilidad estructural facilita el reconocimiento de la enzima, debido a que las enzimas reconocen formas, de modo tal que las polimerasas de ADN solo pueden reconocer el carbono estable sobre el cual puede realizarse el ataque nuclefilico.
Figura
41. ¿Cómo se replica el ADN en el lado inverso a
la síntesis 5→3?. Más que hebra líder deberíamos decirle lado líder,
siempre que un primer o cebador expone una punta 3´, la ADN polimerasa puede
alargarla en la dirección de abertura de la hebra. Dado que la hebra se abre
bidireccionalmente, esto permite que dos lados puedan copiarse continuamente.
El problema es lo que ocurre en la dirección 5´ hacia atrás.
La síntesis de ADN es un proceso que gasta enormes cantidades de energía metabólica. Esto se evidencia en la estructura misma de los nucleótidos adicionados, pues se encuentran en la forma trifosfatada. De forma homóloga a la adenosina que al adquirir tres grupos fosfato se convierte en la molécula de mayor energía en los sistemas biológicos, los nucleótidos del ADN deben estar en una forma trifosfatada para poder ser empleados por la ADN polimerasa. Cuando la polimerasa adiciona un nucleótido a la cadena creciente, debe cortar dos de los grupos fosfato para que la reacción pueda realizarse. La adición del nucleótido depende de su capacidad para ser reconocido por la base complementaria del templado, formando puentes de hidrógeno estables, aunque en ocasiones este mecanismo de reconocimiento falla generando una mutación puntual o SNP.
Figura
42. Replicación semidiscontínua. Para poder copiar lo que hay atrás del extremo 5´
es necesario adicionar un nuevo cebador o primer y seguir copiando desde la
dirección 3´hacia el primer cebador. Esto implica que solo puede copiarse por
segmentos a medida que la hebra se abre por el otro lado, adicionando un cebador a la vez y copiando
parte por parte.
La polimerización de una cadena de ADN requiere de un primer o cebador, la polimerasa de ADN se coloca en el carbono más estable que posee un grupo fosfato, es decir, el carbono 3´. El modelo simplificado más próximo para explicar la adición de nucleótidos a la cadena creciente se denomina mecanismo de doble ión metálico. Y es aquí donde algo interesante emerge, la reacción de polimerización de ADN no puede llevarse a cabo sin la intervención de iones metálicos de magnesio II. En este modelo, uno de los iones de magnesio atrae un protón del carbono 3´, esto desestabiliza al grupo hidroxilo, permitiendo un ataque nuclefilico “el ataque de una molécula cargada negativamente total o parcialmente, de forma tal que atrae electrones de un electrófilo”. El nucleófilo en este caso es el oxígeno 3 del primer grupo fosfato del nucleótido que va a ser adicionado. El segundo magnesio estabiliza el grupo fosfato, promoviendo la liberación de dos de los tres grupos fosfato del nucleótido elegido por la polimeraza. La estabilización se refiere a una neutralización electrostática, los grupos magnesio al tener carga positiva son puentes que evitan que los oxígenos del desoxi-nucleótido se repelan entre sí.
Figura
43. Reiji Okazaki. (1930-1975) fue un biólogo molecular japonés
conocido por sus investigaciones en la replicación del ADN y, especialmente,
por describir el papel de los llamados fragmentos de Okazaki, que descubrió al
trabajar con su esposa Tsuneko Okazaki en 1968.
Como consecuencia de la limitación químico-estructural de las polimerasas de ADN, una de las cadenas crece hacia la pinza de replicación, de forma tal que solo necesita un cebador para copiar. Sin embargo, para la otra hebra, dado que la enzima solo puede copiar en una dirección se crea un dilema bastante interesante. En esta hebra la polimeraza solo podría copias desde la pinza de replicación, lo que dejaría constantemente atrás al cebador, copiando un segmento y dejando al resto de la secuencia que se va abriendo paulatinamente sin copiar. ¿Cómo resolver este dilema estructural? La solución más sencilla sería la evolución de una enzima hipotética que lograra copiar desde la posición 5´, pero nuevamente esa proteína nunca se encontró. La respuesta a esta incógnita es el empleo de un mecanismo relativamente complicado de síntesis a pedazos o discontinua. Es por esto que la replicación del ADN se define como semidiscontinua, una hebra se replica continuamente y la otra discontinuamente.
Figura 44. Tsuneko Okazaki. (1933-viva a 2019) es una científica japonesa conocida por descubrir e investigar los fragmentos de Okazaki, junto con su marido Reiji Okazaki. Los fragmentos de Okazaki han contribuido a explicar la replicación del ADN. La doctora Okazaki ha continuado su relación con la academia, contribuyendo con más avances en la investigación del ADN. Graduada en la Universidad de Nagoya en 1956, Tsuneko Okazaki fue la primera mujer profesora en la universidad japonesa de Nagoya, y actualmente es profesora en el Instituto Médico de la Universidad Fujita. En el año 2000 recibió el Premio L'Oréal-UNESCO a Mujeres en Ciencia, que se concede anualmente a cinco científicas distinguidas, una por cada continente.
La
hebra que puede sintetizarse desde un primer en dirección a la pinza de
replicación es copiada de forma continua y es denominada Hebra Líder. La hebra
que debe ser copiada a fragmentos se denomina Hebra Rezagada. Cabe destacar que
esta nomenclatura no tiene nada que ver con la velocidad de replicación,
estudios posteriores en cinética han demostrado que ambas cadenas se sintetizan
más o menos a la misma velocidad, pero si es cierto que la cadena rezagada
requiere de un mecanismo de replicación mucho más complejo que la cadena líder.
Ahora
bien, ¿Qué hay sobre el problema de huevo-gallina entre los cebadores y las
polimerasas? Recapitulando, in vitro las polimerasas de ADN son las proteínas
que sintetizan el ADN, pero no pueden operar a menos que un pequeño fragmento
de ADN una esté pegado a la hebra relajada. La polimerasa debe poder anclarse
al extremo 3´libre del cebador para poder copiar. Lo cual nos trae a que, ¿cómo
se sintetiza un cebador si las polimerasas de ADN lo necesitarían en primera
instancia para poder formarlo? Los biólogos moleculares insinuaron que in vivo
el proceso era diferente, y de hecho lo es.
Estudios posteriores demostraron que los cebadores in vivo no están
compuestos por ADN sino por ARN. El proceso involucra a una enzima llamada
Primasa de ARN, la cual reconoce orígenes de replicación en la hebra y
sintetiza el cebador de ARN. Luego la ADN polimerasa inicia a copiar desde la
punta 3´de la secuencia de ARN.
El
descubrimiento de que una de las hebras se sintetizas a retazos fue realizada
por el biólogo japonés Reiji Okazaki “1930-1975” y por su esposa Tsuneko
Okazaki “1933-viva” de la Universidad de Nagoya en el año de 1968. Nuevamente,
los experimentos involucraban marcar las cadenas crecientes de ADN con algún tipo
de marcador, que después era medido por un instrumento apropiado. En este caso
se empleó tritio en la timidina, un isótopo del hidrógeno radiactivo. Okazaki
encontró que, si se incubaba por unos segundos una bacteria con timidina
marcada con tritio y se las mataba inmediatamente, el ADN extraído podía verse
como fragmentos cortos de unos 1000 a 2000 nucleótidos de largo. En contraste
si las células se las dejaba crecer por un minute o dos, la mayoría de la
timina marcada con tritio hacía parte de una cadena mucho más larga y continua.
Estos resultados implican que, a gran velocidad, el ADN de la hebra rezagada se
sintetizan en fragmentos que son ensamblados en una hebra única. Estos
fragmentos han sido denominados para la posteridad como los Fragmentos de Okazaki.
Lamentablemente el profesor Renji Okazaki murió a temprana edad debido a cáncer
por la exposición a la radiación por la bomba detonada en su ciudad natal,
Hiroshima.
¿Qué sucede con el cebador de ARN?,
Figura 45. Modelo de la replicación del ADN bacteriano (YouTube).
La replicación de ADN es un proceso
semiconservativo en el cual las dos hebras de la molécula madre se relajan.
Cuando una doble hélice de AADN se relaja esta se desenrosca y expone sus bases
nitrogenadas de forma lineal. Aunque esto causa enroscamientos en el resto de
la molécula, los nudos se resuelven mediante las topoisomerasas de ADN.
Posteriormente cada hebra seguirá una ruta diferente. La hebra líder solo
requiere de un cebador o primer para iniciar la síntesis, este cebador se
sintetiza en un lugar de la secuencia llamado origen de replicación, el cual es
reconocido por una ARN polimerasa, que sintetiza el primer. De esta forma la
ADN polimerasa III se coloca en la punta 3´ del cebador de ARN y comienza a
copiar de forma continua a lo largo de la hebra. La otra hebra debe ser copiada
en fragmentos, por lo que la ARN polimerasas que sintetiza el primer debe
reconocer de corma cíclica varios orígenes de replicación, la ADN polimerasa
III reconoce estos fragmentos t los copia de vez en vez, o en fragmentos
“llamados fragmentos de Okazaki”. Finalmente, los primer de ARN son
reemplazados por una secuencia de ADN por medio de la enzima ADN ligasa.















No hay comentarios:
Publicar un comentario