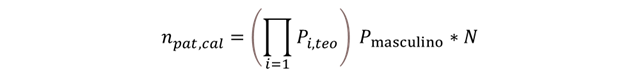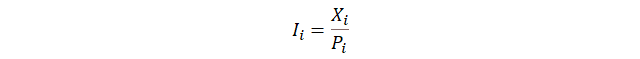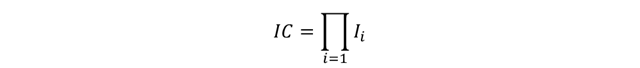(Ciencias de Joseleg) (Biología) (Teoría de la Biología) (Genética moderna) (Introducción) (Naturaleza química del gen) (Conflicto por la estructura del ADN) (Estructura del ADN) (La replicación del ADN) (Síntesis de proteínas) (Denaturación y renaturación) (Estructura del genoma) (Tipos de mutaciones) (Elementos móviles del genoma) (Identificación humana) (Referencias bibliográficas)
Bailey, J. A., Gu, Z., Clark, R. A., Reinert, K., Samonte, R. V, Schwartz, S., … Eichler, E. E. (2002). Recent segmental duplications in the human genome. Science, 297(5583), 1003–1007.
Cardoso-Moreira, M., & Long, M.
(2012). The origin and evolution of new genes. En Evolutionary Genomics
(pp. 161–186). Springer.
Carey, N. (2015). Junk DNA: a
journey through the dark matter of the genome. Columbia University Press.
Carroll, S. B. (2008). Evo-devo and
an expanding evolutionary synthesis: a genetic theory of morphological
evolution. Cell, 134(1), 25–36.
Conant, G. C., & Wolfe, K. H.
(2008). Turning a hobby into a job: how duplicated genes find new functions. Nature
Reviews Genetics, 9(12), 938.
Consortium, E. P. (2012). An
integrated encyclopedia of DNA elements in the human genome. Nature, 489(7414),
57.
Consortium, I. H. G. S. (2001).
Initial sequencing and analysis of the human genome. nature, 409(6822),
860.
Cusanelli, E., & Chartrand, P.
(2014). Telomeric noncoding RNA: telomeric repeat‐containing RNA in telomere
biology. Wiley Interdisciplinary Reviews: RNA, 5(3), 407–419.
Harrison, P. M., Echols, N., &
Gerstein, M. B. (2001). Digging for dead genes: an analysis of the
characteristics of the pseudogene population in the Caenorhabditis elegans
genome. Nucleic acids research, 29(3), 818–830.
Hawkins, J. S., Kim, H., Nason, J.
D., Wing, R. A., & Wendel, J. F. (2006). Differential lineage-specific
amplification of transposable elements is responsible for genome size variation
in Gossypium. Genome research, 16(10), 1252–1261.
Hayashi, Y., Aita, T., Toyota, H., Husimi, Y., Urabe, I.,
& Yomo, T. (2006). Experimental rugged
fitness landscape in protein sequence space. PLoS One, 1(1), e96.
Hooper, J. E. (2014). A survey of
software for genome-wide discovery of differential splicing in RNA-Seq data. Human
genomics, 8(1), 3.
Karp, G. C. (2013). Cell and
Molecular Biology, Concepts and Experiments (7a ed.). USA: Wiley Online
Library.
Kellis, M., Wold, B., Snyder, M. P.,
Bernstein, B. E., Kundaje, A., Marinov, G. K., … Dekker, J. (2014). Defining
functional DNA elements in the human genome. Proceedings of the National
Academy of Sciences, 111(17), 6131–6138.
Klug, W. S., Cummings, M. R.,
Spencer, C. A., & Palladino, M. A. (2012). Concepts of Genetics (10a
ed.). USA: Pearson.
Li, M., Marin-Muller, C., Bharadwaj,
U., Chow, K.-H., Yao, Q., & Chen, C. (2009). MicroRNAs: control and loss of
control in human physiology and disease. World journal of surgery, 33(4),
667–684.
Lieberman, M. A., & Rice, R.
(2014). Biochemistry, Molecular, Biology, and Genetics (6a ed.).
Lippincott Williams & Wilkins.
Lopez, J. V, Yuhki, N., Masuda, R.,
Modi, W., & O’Brien, S. J. (1994). Numt, a recent transfer and tandem
amplification of mitochondrial DNA to the nuclear genome of the domestic cat. Journal
of molecular evolution, 39(2), 174–190.
Maere, S., De Bodt, S., Raes, J.,
Casneuf, T., Van Montagu, M., Kuiper, M., & Van de Peer, Y. (2005).
Modeling gene and genome duplications in eukaryotes. Proceedings of the
National Academy of Sciences, 102(15), 5454–5459.
Maranda, V., Sunstrum, F. G., & Drouin, G. (2019). Both male and female gamete generating cells produce
processed pseudogenes in the human genome. Gene, 684, 70–75.
Marshall, C. R., Raff, E. C., &
Raff, R. A. (1994). Dollo’s law and the death and resurrection of genes. Proceedings
of the National Academy of Sciences, 91(25), 12283–12287.
Morris, K. V. (2012). Non-coding
RNAs and epigenetic regulation of gene expression: Drivers of natural selection.
Horizon Scientific Press.
Nielsen, H., & Johansen, S. D.
(2009). Group I introns: moving in new directions. RNA biology, 6(4),
375–383.
Ohno, S. (1970). Evolution by Gene
Duplication Springer, New York.
Pennisi, E. (2012). ENCODE project
writes eulogy for junk DNA. American Association for the Advancement of
Science.
Petrov, D. A., & Hartl, D. L.
(2000). Pseudogene evolution and natural selection for a compact genome. Journal
of Heredity, 91(3), 221–227.
Qiu, Y., Taylor, A. B., &
McMANUS, H. A. (2012). Evolution of the life cycle in land plants. Journal of
Systematics and Evolution, 50(3), 171–194.
Rouquier, S., Blancher, A., &
Giorgi, D. (2000). The olfactory receptor gene repertoire in primates and
mouse: evidence for reduction of the functional fraction in primates. Proceedings
of the National Academy of Sciences, 97(6), 2870–2874.
Tutar, Y. (2012). Pseudogenes. Comparative
and functional genomics, 2012.
Visel, A., Rubin, E. M., &
Pennacchio, L. A. (2009). Genomic views of distant-acting enhancers. Nature,
461(7261), 199.
Wapinski, I., Pfeffer, A., Friedman,
N., & Regev, A. (2007). Natural history and evolutionary principles of gene
duplication in fungi. Nature, 449(7158), 54.
William, A. D., & Ruse, M.
(2004). Debating design: from Darwin to DNA.
Xie, J., Chen, S., Xu, W., Zhao, Y.,
& Zhang, D. (2019). Origination and Function of Plant Pseudogenes. Plant
signaling & behavior, 14(8), 1625698.
Zhang, F., Zhou, Z., Xu, X., &
Wang, X. (2002). A juvenile coelurosaurian theropod from China indicates
arboreal habits. Naturwissenschaften, 89(9), 394–398.
Zhang, J. (2003). Evolution by gene
duplication: an update. Trends in ecology & evolution, 18(6),
292–298.
Zhang, J., Zhang, Y., &
Rosenberg, H. F. (2002). Adaptive evolution of a duplicated pancreatic
ribonuclease gene in a leaf-eating monkey. Nature genetics, 30(4),
411.
Zheng, D., Frankish, A., Baertsch,
R., Kapranov, P., Reymond, A., Choo, S. W., … Snyder, M. (2007). Pseudogenes in
the ENCODE regions: consensus annotation, analysis of transcription, and
evolution. Genome research, 17(6), 839–851.