(Ciencias de Joseleg) (Biología) (Teoría
de la Biología)
(Genética moderna) (Introducción) (Naturaleza
química del gen) (Conflicto
por la estructura del ADN) (Estructura
del ADN) (La
replicación del ADN) (Síntesis
de proteínas) (Denaturación
y renaturación) (Estructura
del genoma) (Tipos
de mutaciones) (Elementos
móviles del genoma) (Identificación
humana) (Referencias
bibliográficas)
Los estudios sobre la denaturación, la renaturación y sus velocidades permitieron identificar aspectos notables de la organización del genoma de los eucariotas. Cuando los fragmentos del ADN de plantas y animales son recanalizados, la curva típica muestra más o menos tres regiones que corresponde más o menos a tres diferentes tipos de secuencias de ADN. Las tres clases se recanalizan a velocidades diferentes, lo cual depende que tan al azar o que tan repetitiva es la secuencia, a mayor sea la repetitividad, más rápida es la recanalización. Las tres clases se denominan:
1.
Fracción
altamente repetitiva
2.
Fracción
moderadamente repetitiva
3.
Fracción
no repetitiva
En promedio constituyen entre 1% y el 10% de total de pres de bases del ADN. Las secuencias altamente repetitivas son por lo general muy cortas (las más grandes llegan a los cientos de pares de bases). Se encuentran agrupadas en nidos en los cuales una determinada secuencia se repite de forma cíclica persistente “tándem”. Las secuencias altamente repetitivas pueden clasificarse e varias categorías, incluyendo: ADN satélite, minisatélite, microsatélite. Estos nombres se originan a partir de propiedades físicas.
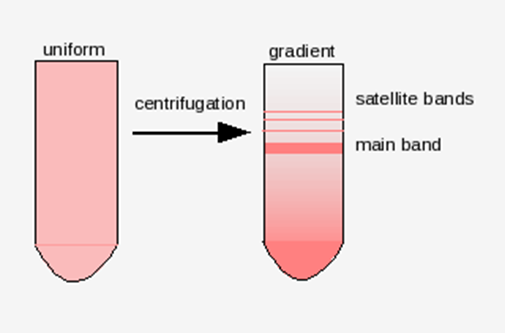
Figura
58. Los satélites al ADN principal. Cuando se centrifuga el ADN se forman varias
bandas, una es la principal que es más densa, mientras que otras son más
pequeñas y fueron denominadas satélites.
En
promedio constituyen entre 1% y el 10% de total de pres de bases del ADN. Las
secuencias altamente repetitivas son por lo general muy cortas (las más grandes
llegan a los cientos de pares de bases). Se encuentran agrupadas en nidos en
los cuales una determinada secuencia se repite de forma cíclica persistente
“tándem”. Las secuencias altamente repetitivas pueden clasificarse e varias
categorías, incluyendo: ADN satélite, minisatélite, microsatélite. Estos
nombres se originan a partir de propiedades físicas.
El ADN
satélite consiste en repeticiones entre cinco y algunos cientos de pares de
bases, los cuales forman nidos “clusters” extremadamente largos, cada uno
conteniendo millones de pares de bases. El nombre satélite emergió de una
propiedad física que emerge de la alta repetición de estas secuencias en
algunas especies.
La
composición entre el satélite y el ADN no repetitivo es tan diferente que al
ser centrifugado los fragmentos se separan en bandas diferentes, el ADN
satélite se ubica en una banda satélite con respecto al resto de gradiente de
centrifugación. El ADN satélite tiende a acumular mutaciones rápidamente, lo
que causa de las secuencias homólogas en una secuencia satélite cambien mucho
aun en especies altamente relacionadas. Lo anterior permite realizar procesos
taxonómicos en especies muy relacionadas.
Las
secuencias minisatélite rondan repeticiones entre 10 y 100 pares de bases
agrupadas en nidos “clusters” de unas 3000 repeticiones. Por lo anterior, las
secuencias minisatélite ocupan regiones del ADN considerablemente más pequeñas
que las secuencias satélites, sin embargo, los minisatélites tienden a ser
inestables, lo cual hace que varíen de una generación a la siguiente. En
consecuencia, la longitud de un
Los ADN
microsatélite son las secuencias más cortas de todas las pertenecientes a la
fracción altamente repetitiva. Típicamente son fragmentos de 1 a 9 pares de
bases, y sus nidos “clusters” van de 10 a 40 repeticiones, los cuales se
encuentran dispersos a través del genoma. Las enzimas especializadas en copiar
al ADN tienen muchos problemas en copiar secuencias altamente repetitivas, lo
cual conlleva a una tasa de mutaciones relativamente alta. Lo anterior implica
que la longitud de un determinado microsatélite puede variar entre especies,
variedades, e incluso hasta miembros de una misma familia. Dado lo anterior,
los microsatélites se han convertido en la principal herramienta para el
análisis de las poblaciones y las pruebas de identificación humana. El ADN
microsatélite experimenta una variación lo suficientemente rápida como para
poder realizar un análisis genealógico de poblaciones al interior de una misma
especie, e incluso, algunos marcadores son tan mutables como para poder
diferenciar hasta individuos al interior de una misma población. Otros estudios
ya habían planteado la plausibilidad de que todos los humanos desandemos de una
pequeña población africana que vivió hace unos 150 000-250 000 años.
En base
a esto, es posible afirmar que las poblaciones africanas han tenido más tiempo
para diversificarse genéticamente, mientras que solo una fracción decidió
emigrar al norte y dar lugar a todas las demás poblaciones del mundo. Lo
anterior implicaría que todas las poblaciones del planeta poseen solo una
fracción de la diversidad de las poblaciones africanas. Otra conclusión algo paradójica es que, un
africano puede llegar a parecerse genéticamente más a un europeo “en el caso
que dicho africano pertenezca al pool genético del cual descienden los demás
seres humanos del planeta” que con el vecino de una tribu cercana. En cualquier
caso, estas conclusiones se basan en estudios cuantitativos realizados a través
de marcadores como los minisatelites y microsatélites. En cualquier caso, desde
la ciencia y las teorías evolutivas modernas, solo existe una raza de una sola
especie humana viviendo en la actualidad, lo único que debe ser analizado
cuantitativamente como un grupo cerrado relativamente es una población, pero
las poblaciones pueden estar compuestas por una alta diversidad de grupos
étnicos.
La mayor
parte de los
Las
secuencias moderadamente repetitivas en las plantas y los animales pueden
variar desde el 20% al 80% del genoma total de la especie. Esta fracción
incluye secuencias que se repiten a través del genoma en cualquier parte desde
unas pocas a unas decenas de miles de veces. Se incluyen en la fracción
moderadamente repetitiva algunas secuencias que están involucradas con la
expresión de ciertos genes, ya sea en términos de proteínas o ARN, así como
aquellas que carecen de funciones codificantes, sea porque están involucradas
en la regulación o porque en verdad no poseen función alguna.
Esta
fracción incluye al ADN de genes que codifican para ARNs así como para ciertas
proteínas regulatorias y estructurales importantes como las histonas. Las
secuencias repetitivas que codifican para estos productos tándem a ser
repetitivas y organizadas en tándem. Los genes que codifican para ARNs como los
que componen al ribosoma deben estar repetidos múltiples veces debido a que
estos no son amplificados posteriormente por el ribosoma. Aunque la producción
de histonas si requiere de la amplificación de los ribosomas, se requiere
muchísimas copias de estas proteínas durante el inicio del ciclo celular, que
múltiples templados para su producción deben estar presentes de forma
simultánea.
La
mayor parte de las secuencias moderadamente repetitivas no poseen una función
codificante, es decir, es incapaz de generar un producto génico sea ARN o
proteínas debido a que no puede ser leído por las enzimas especializadas. A
diferencia de las secuencias altamente repetitivas, las secuencias moderadamente
repetitivas no se encuentran alojadas en nidos en tándem, por el contrario, se
encuentran dispersas en toda la extensión del genoma. Esta fracción es dividida
en dos grupos bastante famosos. El primer grupo se denomina SINE que significa
elementos cortos intercalados “short interspersed elements” y el segundo grupo se denomina
LINE que significa elementos largos intercalados “long interspersed elements”.
Como
fue predicho por los mendelianos en los estudios sobre la genética clásica los
genes se encuentran ubicados en una sola copia por cada segmento de un
cromosoma simple. Usando una célula eucariota se encuentra en este estado se la
denomina haploide, y por lo general corresponde a los gametos sexuales
reproductivos. Los genes por lo general son secuencias no repetitivas, aunque
existen excepciones, sin embargo, es posible afirmar que entre más lento es la
recanalización de las dos hebras durante la renaturación menos repetitivo es el
ADN. En ocasiones la recanalización es tan lenta en estas fracciones de ADN que
puede presumirse que dichas secciones no son repetitivas en absoluto, en otras
palabras, que los genes se encuentran en copias únicas en ciertas ocasiones. Estudios
posteriores demostraron que los genes que provocaban la lentitud de la
recanalización se comportaban de forma mendeliana, lo cual de por sí ya es una
rareza.
La definición de gen y
en una fracción de ADN no repetitiva
Estos
genes que se encuentran en copias únicas y se comportan de forma mendeliana
clásica nos permiten ingresar el concepto de gen y locus, por lo menos en sus definiciones más básicas. Un gen es
una secuencia de ADN que almacena la información genética necesaria para la producción
de otras moléculas como el ARN y las proteínas. El
Existen
genes que, si se presentan en copias de sí mismos, y generan o herencias no
mendelianas o rasgos diferentes o acoplados con sigo mismos. Estos genes
copiados debido a su naturaleza misma pueden agruparse en un mismo grupo que
posee una estructura muy similar. A estos grupos se los ha denominado familias
de genes.
Virtualmente
todos los genes existentes pueden ser agrupados en familias de genes, esto se
debe a que los genes se producen más fácilmente de genes previos con leves
modificaciones, que a partir de secuencias no codificantes. Estos últimos genes
se denominan genes que han evolucionado de Novo,
o lo que en términos pragmáticamente estadísticos podríamos decir, es como si
evolucionaran de la nada o del caos.
Sin
embargo, en cuanto a las familias de genes, existen algunas más famosas que
otras, especialmente por presentarse copias al interior de un mismo genoma,
este es el caso de las globinas, las actinas, las miosinas, los colágenos, las
tubulinas, las integrinas, las proteasas de serina entre casi todas las demás
proteínas eucariotas.
Ahora
que el genoma humano ha sido secuenciado y después de más de una década de
análisis es posible afirmar que la fracción de ADN codificante es
increíblemente pequeña en comparación con el resto del genoma. De hecho, si
esto se le hubiera sugerido a un genetista de la década de 1960, él hubiera
considerado tal propuesta como ridícula. ¿Dada nuestra enorme complejidad cómo
es posible que menos del 1,5% de nuestro genoma sea empleado para nuestra
configuración corporal? Y aun así esa es la realidad que ha emergido de los
estudios más modernos sobre la biología molecular y la genética.
Las
secuencias de ADN no codificantes son componentes del ADN de un organismo que
no codifican secuencias de proteínas. Aunque se podría pensar que se trata de
ADN satélite y microsatélite la verdad es un poco más complicada, parte del ADN
no codificante se transcribe en moléculas de ARN no codificantes funcionales
(por ejemplo, ARN de transferencia, ARN ribosómico y ARN reguladores). Otras
funciones del ADN no codificante incluyen la regulación transcripcional y
traduccional de secuencias codificantes de proteínas, regiones de unión de
andamios, orígenes de replicación de ADN, centrómeros y telómeros.
La
cantidad de ADN no codificante varía mucho entre especies. A menudo, solo un
pequeño porcentaje del genoma es responsable de codificar las proteínas, pero
se muestra que un porcentaje creciente tiene funciones reguladoras. Cuando hay
mucho ADN no codificante, una gran proporción parece no tener función
biológica, como se predijo en la década de 1960. Desde entonces, esta porción
no funcional “al menos para su individuo portador” ha sido controvertidamente
llamada "ADN basura" (Pennisi, 2012).
El
proyecto internacional Encyclopedia
of DNA Elements
(ENCODE) descubrió, mediante enfoques bioquímicos directos, que al menos el 80%
del ADN genómico humano tiene actividad bioquímica (E. P. Consortium, 2012). Aunque esto no fue necesariamente inesperado
debido a décadas anteriores de investigación que descubrieron muchas regiones
no codificantes funcionales (Carey, 2015), algunos científicos criticaron
la conclusión por combinar la actividad bioquímica con la función biológica,
pues aunque se generen trascritos de ARN mensajero, muchos de estos no sirven
para nada, siendo pseudogenes degenerados por mutaciones deletéreas (Maranda, Sunstrum, &
Drouin, 2019; Xie, Chen, Xu, Zhao, & Zhang, 2019).
Las
estimaciones para la fracción biológicamente funcional del genoma humano
basadas en genómica comparativa oscilan entre 8 y 15% (Kellis et al., 2014). Sin embargo, otros han
argumentado en contra de depender únicamente de estimaciones de genómica
comparativa debido a su alcance limitado. Se ha descubierto que el ADN no
codificante está involucrado en la actividad epigenética y redes complejas de
interacciones genéticas y se está explorando en la biología evolutiva del
desarrollo (Morris, 2012).
En la actualidad se
distinguen las siguientes categorías de ADN no codificante.
Los ARN
no codificantes son moléculas de ARN funcionales que no se traducen en
proteínas. Los ejemplos de ARN no codificante incluyen ARN ribosómico, ARN de
transferencia, ARN que interactúa con Piwi y los microARN. Se predice que los
microARN controlan la actividad traduccional de aproximadamente el 30% de todos
los genes que codifican proteínas en mamíferos y pueden ser componentes vitales
en la progresión o el tratamiento de diversas enfermedades, incluido el cáncer,
las enfermedades cardiovasculares y la respuesta del sistema inmunitario a la
infección (Li et al., 2009).
Los elementos
reguladores de cis son secuencias que controlan la transcripción de un gen
cercano. Muchos de estos elementos están involucrados en la evolución y el
control del desarrollo (Carroll, 2008). Los elementos cis pueden estar ubicados en regiones no
traducidas de 5 'o 3' o dentro de intrones. Los elementos transreguladores controlan la transcripción
de un gen distante. Los promotores facilitan la transcripción de un gen
particular y están típicamente aguas arriba de la región de codificación. Las
secuencias potenciadoras también pueden ejercer efectos muy distantes sobre los
niveles de transcripción de los genes (Visel, Rubin, &
Pennacchio, 2009).
Los
intrones son secciones no codificantes de un gen, transcritas en la secuencia
precursora de ARNm, pero finalmente eliminadas por empalme de ARN durante el
procesamiento para madurar ARN mensajero. Muchos intrones parecen ser elementos
genéticos móviles(Nielsen & Johansen, 2009). Los estudios de intrones del
grupo I de los protozoos de Tetrahymena
indican que algunos intrones parecen ser elementos genéticos egoístas,
neutrales para el huésped porque se eliminan de los exones flanqueantes durante
el procesamiento del ARN y no producen un sesgo de expresión entre alelos con y
sin el intrón (Nielsen & Johansen, 2009). Algunos intrones parecen tener
una función biológica significativa, posiblemente a través de la funcionalidad
de la ribozima que puede regular la actividad de tRNA y rRNA, así como la
expresión de genes que codifican proteínas, evidente en los huéspedes que se
han vuelto dependientes de tales intrones durante largos períodos de tiempo;
Por ejemplo, el intrón trnL se encuentra en todas las plantas verdes y parece
haber sido heredado verticalmente durante varios miles de millones de años,
incluidos más de mil millones de años dentro de los cloroplastos y otros 2-3
billones de años antes en los ancestros cianobacterianos de los cloroplastos (Nielsen & Johansen, 2009). Los intrones tambien son
importantes para almacenar más de un gen en un mismo locus, por medio del
proceso de maduración diferencial de un ARNm inmaduro (Hooper, 2014).
Los
pseudogenes son secuencias de ADN, relacionadas con genes conocidos, que han
perdido su capacidad de codificación de proteínas o ya no se expresan en la
célula. Los pseudogenes surgen de la retrotransposición o la duplicación
genómica de genes funcionales, y se convierten en "fósiles genómicos"
que no funcionan debido a mutaciones que impiden la transcripción del gen, como
dentro de la región promotora del gen, o alteran fatalmente la traducción del
gen, como codones de parada prematura o cambios de marco de lectura (Zheng et al., 2007). Los pseudogenes que resultan
de la retrotransposición de un ARN intermedio se conocen como pseudogenes
procesados; Los pseudogenes que surgen de los restos genómicos de genes
duplicados o residuos de genes inactivados son pseudogenes no procesados (Zheng et al., 2007). Las transposiciones de genes
mitocondriales que alguna vez fueron funcionales desde el citoplasma al núcleo,
también conocidos como NUMT, también califican como un tipo de pseudogen común (Lopez, Yuhki, Masuda, Modi,
& O’Brien, 1994). Los números ocurren en muchos
taxones eucariotas. Si bien la Ley de Dollo sugiere que la pérdida de función
en los pseudogenes es probablemente permanente, los genes silenciados en
realidad pueden conservar la función durante varios millones de años y pueden
"reactivarse" en secuencias codificantes de proteínas (Marshall, Raff, & Raff,
1994) y se transcribe activamente un número sustancial de
pseudogenes (Tutar, 2012).
Debido a que se presume que los pseudogenes cambian sin restricción evolutiva,
pueden servir como un modelo útil del tipo y las frecuencias de varias
mutaciones genéticas espontáneas (Petrov & Hartl, 2000).
Los transposones y
retrotransposones son elementos genéticos móviles. Las secuencias repetidas de
retrotransposón, que incluyen elementos nucleares intercalados largos (LINE) y
elementos nucleares intercalados cortos (SINE), representan una gran proporción
de las secuencias genómicas en muchas especies. Las secuencias Alu,
clasificadas como un elemento nuclear corto intercalado, son los elementos
móviles más abundantes en el genoma humano. Se han encontrado algunos ejemplos
de SINE que ejercen control transcripcional de algunos genes que codifican
proteínas.
Las secuencias endógenas
de retrovirus son el producto de la transcripción inversa de genomas de
retrovirus en genomas de células germinales. La mutación dentro de estas
secuencias retro-transcritas puede inactivar el genoma viral. Más del 8% del
genoma humano está formado por secuencias de retrovirus endógenas (en su
mayoría descompuestas), como parte de la fracción de más del 42% que se deriva
de los retrotransposones, mientras que otro 3% puede identificarse como restos
de transposones de ADN. Se espera que gran parte de la mitad restante del
genoma que actualmente no tiene un origen explicado haya encontrado su origen
en elementos transponibles que estuvieron activos hace tanto tiempo (> 200
millones de años) que las mutaciones aleatorias los han vuelto irreconocibles (I. H. G. S. Consortium, 2001). La variación del tamaño del genoma en al menos dos tipos de
plantas es principalmente el resultado de secuencias de retrotransposones (Hawkins, Kim, Nason, Wing,
& Wendel, 2006).
Los telómeros son regiones de ADN repetitivo al final de un cromosoma, que proporcionan protección contra el deterioro cromosómico durante la replicación del ADN. Estudios recientes han demostrado que los telómeros funcionan para ayudar en su propia estabilidad. El ARN que contiene repetición telomérica (TERRA) son transcripciones derivadas de los telómeros. Se ha demostrado que TERRA mantiene la actividad de la telomerasa y alarga los extremos de los cromosomas (Cusanelli & Chartrand, 2014).

No hay comentarios:
Publicar un comentario